尊敬的国家知识产权局:
您好!我们代表申请人,就国家知识产权局对我们发明专利申请的非正常申请提出的意见进行陈述。本发明旨在提供一种抗生素使用强度动态监测方法及系统,以解决医疗机构抗生素使用实时监控,促进抗生素合理使用的问题。我们对国知局指出的非正常申请提出以下观点:
一、申请人、发明人实际研发能力
茂名市人民医院是一所具有丰富经验和技术力量的医疗机构,其设备先进、科研实力雄厚。医院鼓励并支持医务人员积极申请发明专利,保护其在医疗技术创新方面的成果。发明人胡林辉副主任医师,拥有丰富的医疗经验和硕士学位,已发表多篇高水平SCI论文,具备扎实的研究背景。医院注重知识产权管理,建立了健全的制度确保科研成果的合法性、有效性和可持续发展。我们确信申请人不存在非正常申请的情况。
二、发明人科研成果及研发能力
发明人胡林辉在硕士阶段获得国家奖学金,并在医学领域发表多篇高水平SCI论文。他具有广泛的学科背景,掌握多种统计语言和编程技术。在发明专利的申请和授权方面,发明人胡林辉已申请并获得多项实用新型专利和软件著作权。此外,研发团队核心成员均具有硕士学位,专业背景广泛,为项目提供了全面的支持。
三、研发过程及系统成果
我们在临床需求沟通过程中充分考虑了医院在国家公立医院考核中对抗生素使用指标的监控,经过反复讨论和调试,最终完成了具有实际应用价值的抗生素使用强度动态监测系统。该系统经历了核心功能版的1.0版和后续版本的迭代,以满足各科室的具体管理需求。系统使用效果良好,得到了医务人员的认可。
我们对国家知识产权局关于非正常申请的意见表示理解,但我们坚信我们的申请是经过正常流程、在医疗领域具有实际应用价值的发明专利。我们诚挚希望国家知识产权局能充分考虑我们在陈述书中提出的观点,认可我们的申请是正常、合理、有实质性内容的。我们已经按照国家知识产权局的要求提供了详尽的材料和解释,希望国家知识产权局能够作出转为正常申请的裁定。
最后,我们感谢国家知识产权局对我们专利申请的审查工作,期待国家知识产权局能够根据我们的意见和提供的材料作出合理的裁定。谢谢!
此致,
发明人代表
胡林辉
以下是我们陈述意见的详情,敬请详阅。
1 背景
本发明旨在提供一种抗生素使用强度动态监测方法及系统,以解决医疗机构抗生素使用实时监控,促进抗生素合理使用的问题。该方法能快速、准确、实时地计算全院抗生素使用强度,为医疗机构的相关监测工作提供重要依据考。与传统人工计算的滞后性相比,该系统能实现动态监测,及时发现超标情况并进行调整,具有重要的应用前景。本技术经我们检索,国内外没有先例,经向专利事务所咨询后,具有申请发明专利的前景,具有保护价值后,经医院正规申请流程,经审核同时委托专利事务所申报国家发明专利。申请人不存在非正常申请事实,发明人具有相应研发能力,特向国知局陈述。
2 申请人、发明人实际研发能力
2.1 申请人简介
茂名市人民医院是一所具有悠久历史和丰富经验的综合性医疗机构,医院设备先进、技术力量雄厚。医院拥有2500张病床和3069名在职员工,其中包括466名高级职称人员和303名博士、硕士研究生。医院下设水东湾分院和医院总部,占地面积约450亩,是广东省临床重点专科医院,拥有多个国家专科治疗中心和省级重点专科,并在医疗业务方面取得显著成绩(图 1)。同时,医院积极发展前沿医疗技术,建设高水平临床科研平台(图 2),集聚拔尖医学人才,打造一流医学学科,建立现代医院管理制度,在疫情防控和医疗服务上表现出色,为当地乃至粤西地区的医疗健康事业做出了突出贡献。
在发明专利的申请和授权方面,医院不断加强知识产权的保护意识,推动科研成果的转化和商业化。医院鼓励医务人员积极申请发明专利,保护其在医疗技术创新方面的成果(图 3)。同时,医院还积极与相关机构和企业进行合作,推动科研成果的产业化和市场化。茂名市人民医院也注重知识产权的管理和运用,建立了健全的知识产权管理制度,确保科研成果的合法性、有效性和可持续发展。医院重视发明专利的运营和管理,充分发挥发明专利在技术创新、医疗服务和经济效益等方面的作用。
茂名市人民医院高水平医院建设情况汇报_05.png)
茂名市人民医院高水平医院建设情况汇报_25.png)
茂名市人民医院高水平医院建设情况汇报_30.png)
2.2 发明人履历
| 姓名 | 胡林辉 | 性 别 | 男 | 专业技术职称 | 副主任医师 |  |
||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 出生年月 | 1985.11 | 定 职 时 间 | 2022.12 | |||||||||||||||
| 本专业学会任职 | 广东省器官医学与技术学会重症医学专业委员会委员、广东省医师协会休克专业委员会委员 | |||||||||||||||||
| 电子邮箱 | hulinhui@live.cn | 手机号码 | 13580013426 | 是否归国人员 | 否 | |||||||||||||
| 最后学历 | 硕士研究生 | 毕业时间 | 2019年6月21日 | 毕业院校 | 华南理工大学 | |||||||||||||
| 最高学位 | 硕士 | 学位授予时间 | 2019年6月21日 | 学位授予院校 | 华南理工大学 | |||||||||||||
| 主 要 经 历 |
起止年月 | 工 作 部 门 | 任 职 | |||||||||||||||
| 2020.02至今 | 茂名市人民医院重症医学科三区、科研中心 | 副主任 | ||||||||||||||||
| 2016.09-2019.06 | 华南理工大学、广东省人民医院 | 硕士研究生 | ||||||||||||||||
| 2009.06-2016.08 | 高州市人民医院重症医学科 | 执业医师 | ||||||||||||||||
2.3 发明人科研成果
| 成果(科研论文、教材等)名称 | 发表刊物、出版单位等 | 级别 | 时间 (例202108) |
本人 排 序 |
|---|---|---|---|---|
| A protein encoded by circular ZNF609 RNA induces acute kidney injury by activating AKT/mTOR-autophagy pathway | Molecular Therapy | 1区 Top 期刊 |
202306 | 共同第一作者 |
| Serum N-terminal pro-B-type natriuretic peptide and cystatin C for acute kidney injury detection in critically ill adults in China: a prospective, observational study | BMJ Open | 3区 | 202301 | 共同第一作者 |
| Effects of Juhongtanke oral solution on alleviating the symptoms of community-acquired pneumonia: A multicenter, prospective, randomized controlled trial | Frontiers in Pharmacology | 2区 Top 期刊 |
202210 | 共同第一作者 |
| Urinary proteome analysis ofacute kidney injury inpost-cardiac surgery patientsusing enrichment materials withhigh-resolution massspectrometry | Frontiers in Bioengineering and Biotechnology | 2区 | 202209 | 共同第一作者 |
| A novel strategy sequentially linking mechanical cardiopulmonary resuscitation with extracorporeal cardiopulmonary resuscitation optimizes prognosis of refractory cardiac arrest: an illustrative case series | European Journal of Medical Research | 4区 | 202205 | 第一作者 |
| Variations of urianry N-acetyl-β-D-glucosaminidase levels and its performance in detecting acute kindey injury under different thyroid hoemoness levels: A prospectively-recruited, observtional study | BMJ Open | 3区 | 202203 | 共同第一作者 |
| The incidence, risk factors and outcomes of acute kidney injury in critically ill patients undergoing emergency surgery: a prospective observational study | BMC Nephrology | 4区 | 202201 | 第一作者 |
| Development and Validation of a Nomogram Incorporating Colloid Osmotic Pressure for Predicting Mortality in Critically Ill Neurological Patients | Frontiers in Medicine | 3区 | 202112 | 共同第一作者 |
| C-reactive protein concentration as a risk predictor of mortality in intensive care unit: a multicenter, prospective, observational study | BMC Anesthesiology | 4区 | 202011 | 共同第一作者 |
| 一种鼻饲管 | 实用新型专利 | 202207 | 2 | |
| ANTI-PROTEIN ADSORPTION NASOINTESTINAL TUBE AND PREPARATION METHOD | 实用新型专利 | 202110 | 3 | |
| CT俯卧位乳房检查支撑架 | 实用新型专利 | 202107 | 9 | |
| 硫辛酸多中心研究在线CRF平台1.0 | 软件著作权 | 202204 | 1 | |
| 多中心eCRF平台1.0 | 软件著作权 | 202111 | 1 |
2.3.1 近3年承接的各级科研项目
| 序号 | 项目名称 | 项目来源 及项目编号 |
项目 类型 |
起讫时间 | 资助 经费 |
本人署名 次 序 |
|---|---|---|---|---|---|---|
| 1 | 化橘红制剂缓解新型冠状病毒感染的肺炎症状的随机对照研究 | 茂名市应急科技计划项目 2020YJ01 |
一般/面上项目 | 2020/2/1-2023/2/28 | 20万 | 主持人 |
| 2 | 硫辛酸注射液治疗脓毒症和脓毒性休克成人患者的前瞻性、单盲、随机、安慰剂对照的临床研究 | 茂名市科技专项资金项目-医学科技创新专题 2021KJZXZJYX003 |
一般/面上项目 | 2022/3/1-2024/2/28 | 5万元 | 主持人 |
| 3 | 机械胸外按压序贯体外心肺复苏(ECPR)治疗难治性心跳骤停的临床研究 | 广东省医学科学技术研究基金项目 B2022246 |
一般/面上项目 | 2022/7/1-2024/6/30 | 立项非资助 | 主持人 |
2.3.2 研发能力
发明人本科期间是学术计算机协会成员,最早学习了Foxpro和VBA等编程语言,掌握了基本的编程技术。在研究生阶段系统学习并掌握了统计语言,包括R、SAS和Python等。同时, 为了满足课题组的需求,自学完成了前端编程语言,如HTML、JavaScript和CSS,以及后端语言,比如PHP等。在工作和科研中,为提高效率,开始开发交班程序、数据录入平台等软件平台,在研究生期间跟随导师出色完成相关系列临床研究,发表3篇SCI论文,获得国家奖学金(图 4),并且部分已申请了软件著作权。至今所有软件著作权申请均有效,没有出现申请无效的情况。
(1).jpg)
研发团队核心成员均具有硕士学位,专业背景包含临床医学、流行病学与统计专业,具有感知临床需求及将临床需求转化为数据模型的能力(图 5,图 6,图 7,图 8,图 9,图 10)。






除此之外,成员黄健、梁诚、陈火华分别是我院主管副院长、计算机中心负责人和工程师,为数据接口提供了坚强的协调和技术保障。
团队具有开发软件的成熟经验,成功申请多个软件著作权(图 11,图 12,图 13),锻炼了成熟的数据处理和平台开发能力。



3 研发过程
3.1 临床需求沟通过程
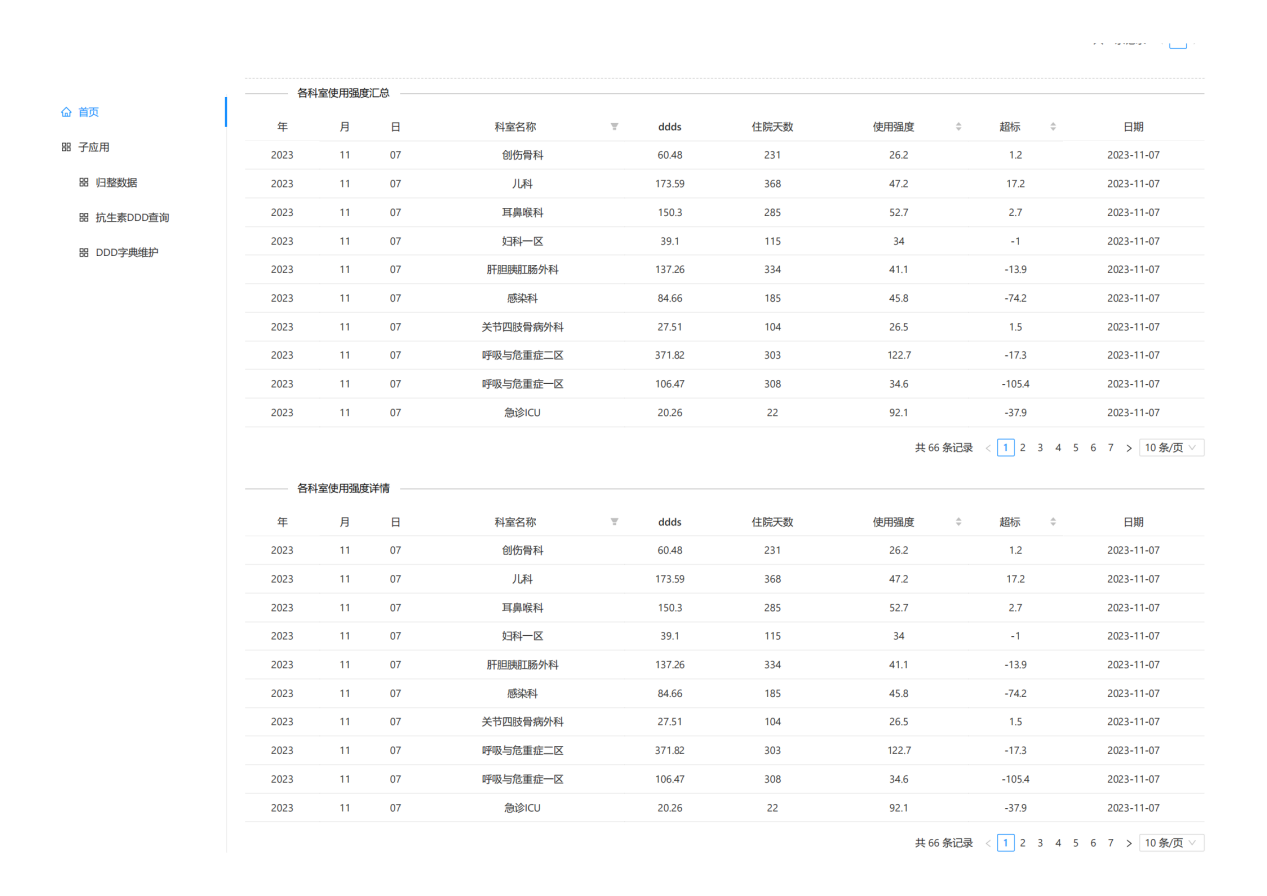
该技术需求来源于医院在国家公立医院考核中对抗生素使用指标的监控,首先是在发明人因科室需要,在科室层面使用。业务院长查房后发现系统不但对科室使用有用,对全院抗生素使用强度监控也具有重要作用,故发明人在计算机科、药剂科的协助下研发出该抗生素使用强度监测系统,对院级及全院60余个科室的抗生素使用强度进行实时监控和管理(图 14,图 15,图 16,图 17)。
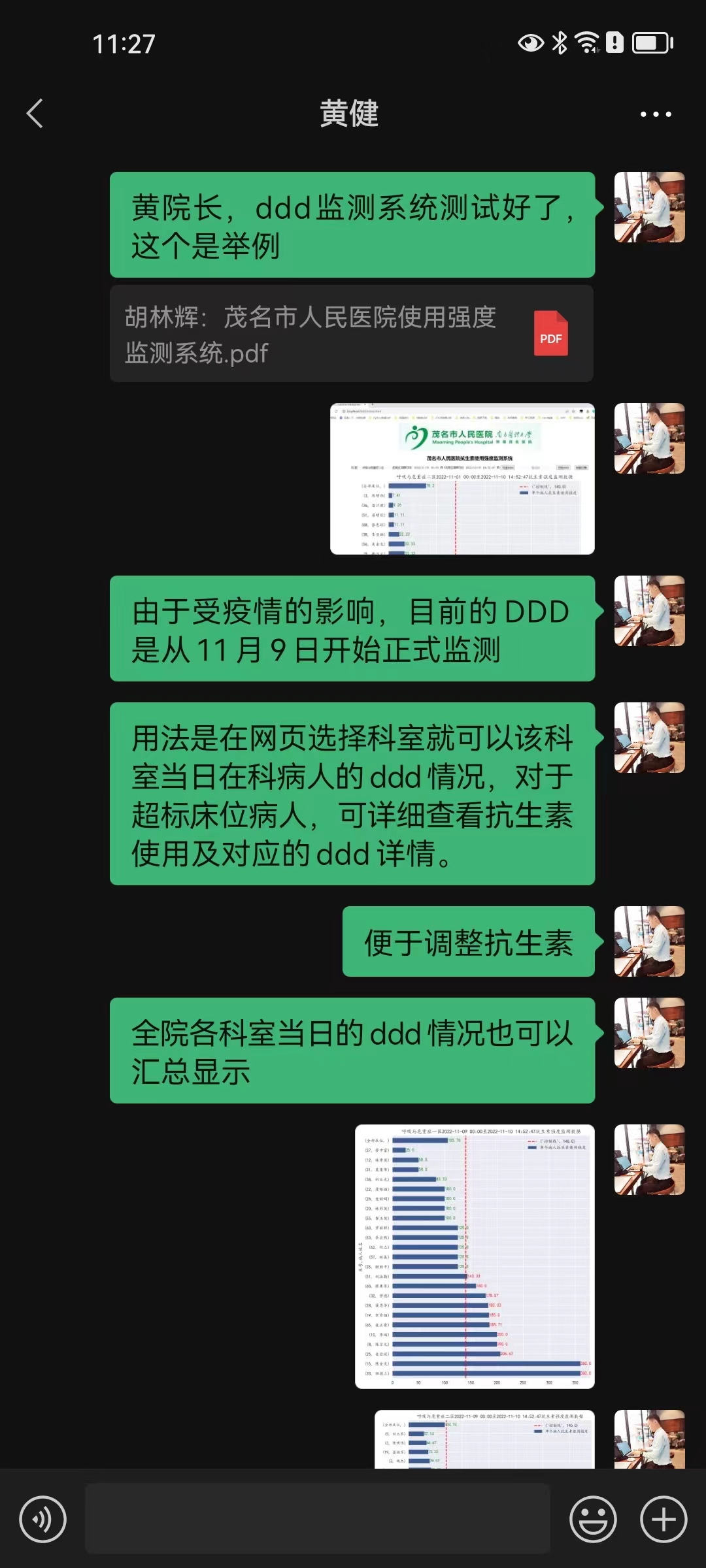
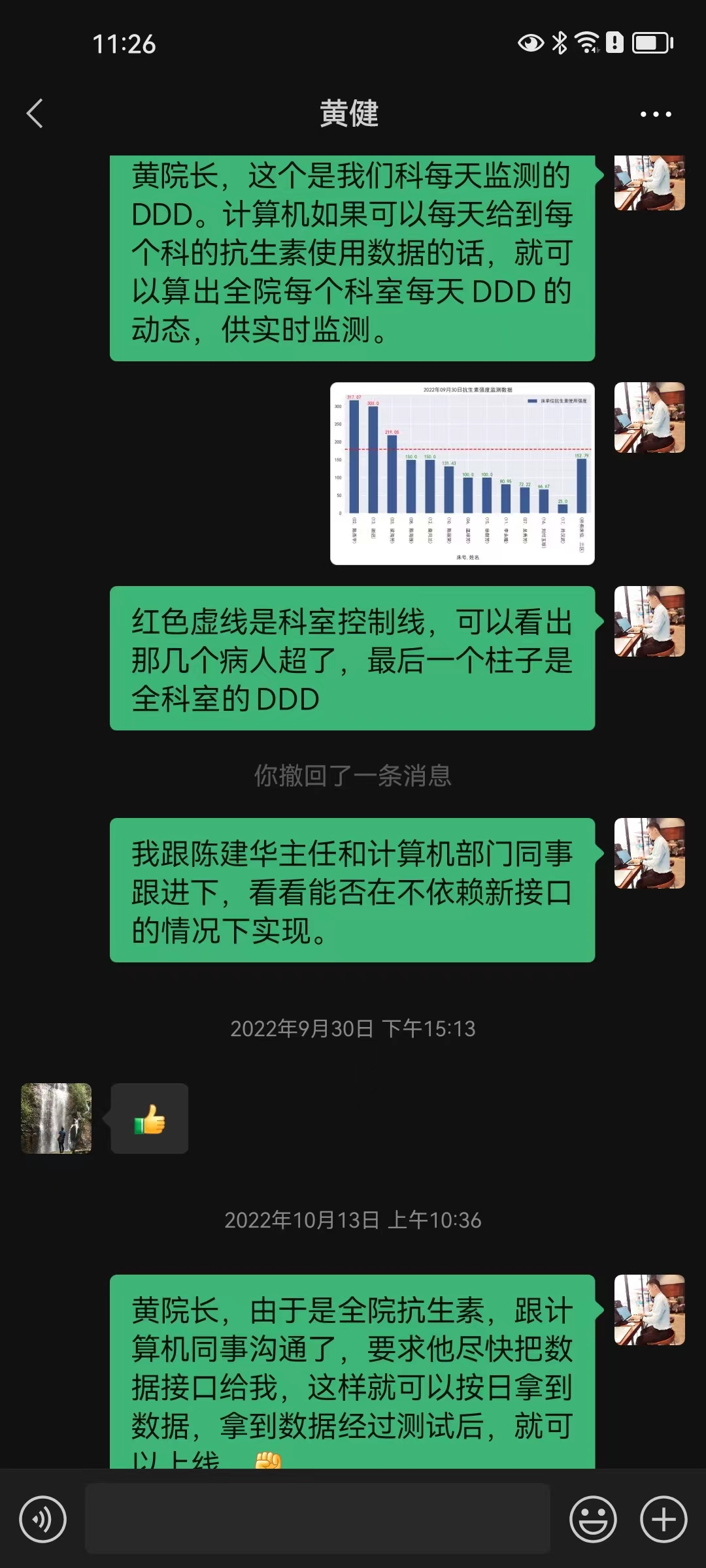
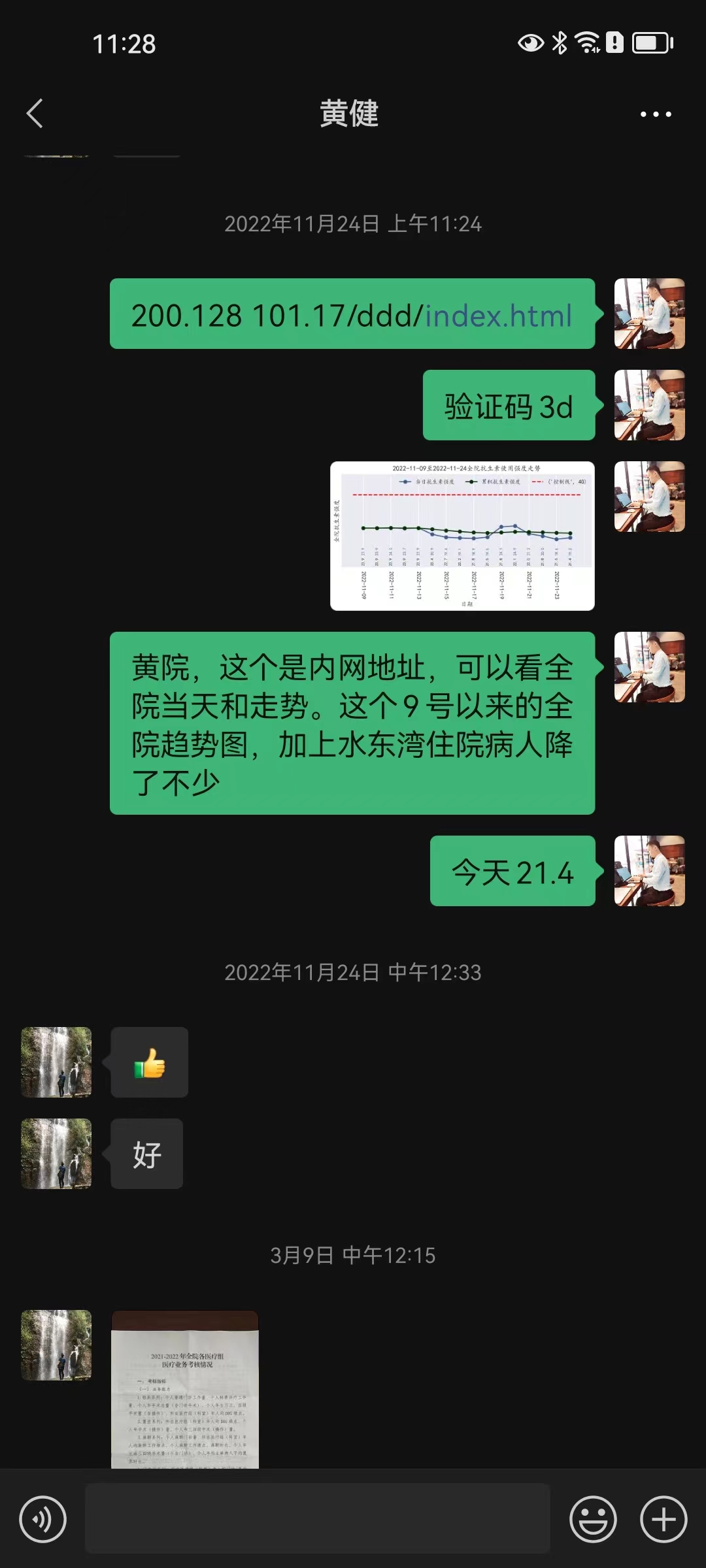
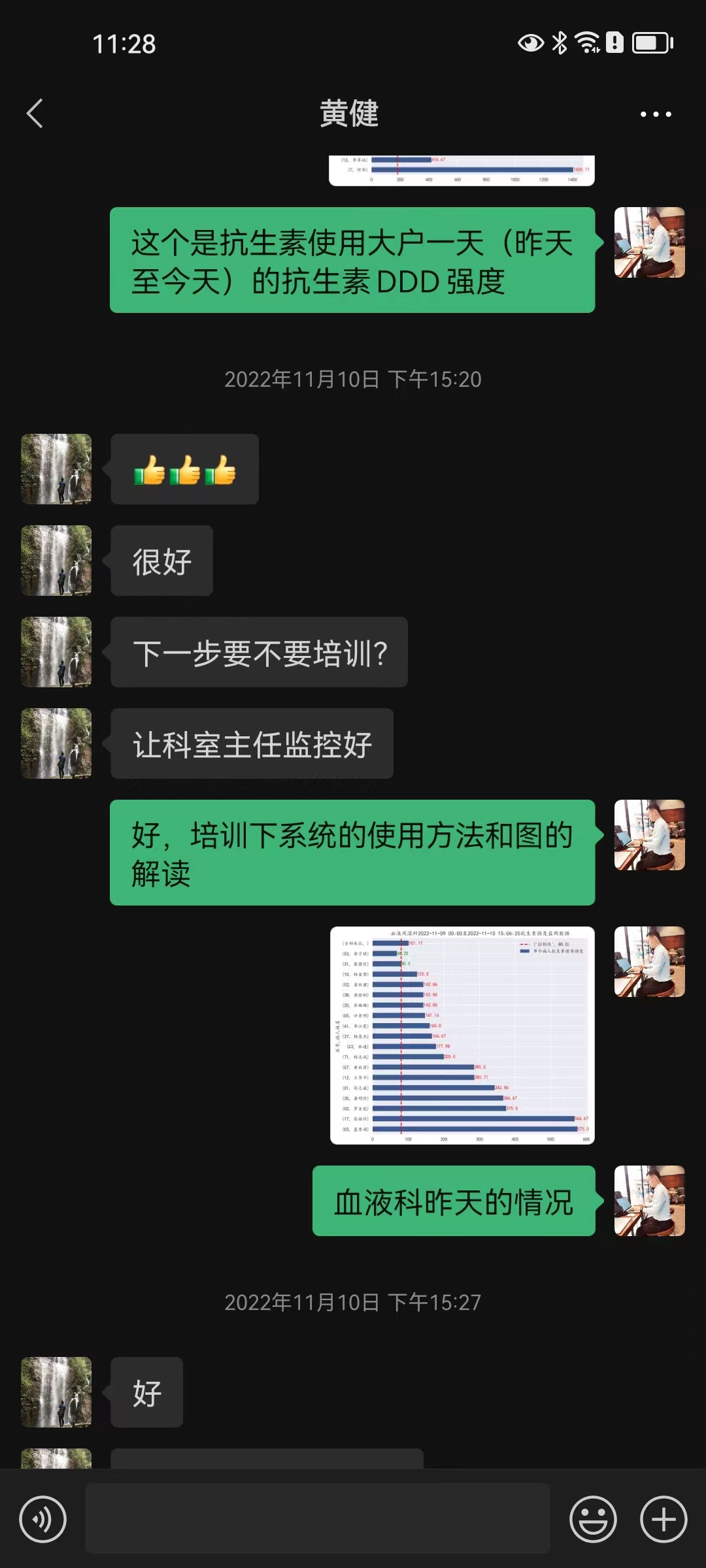
3.2 系统使用界面预览
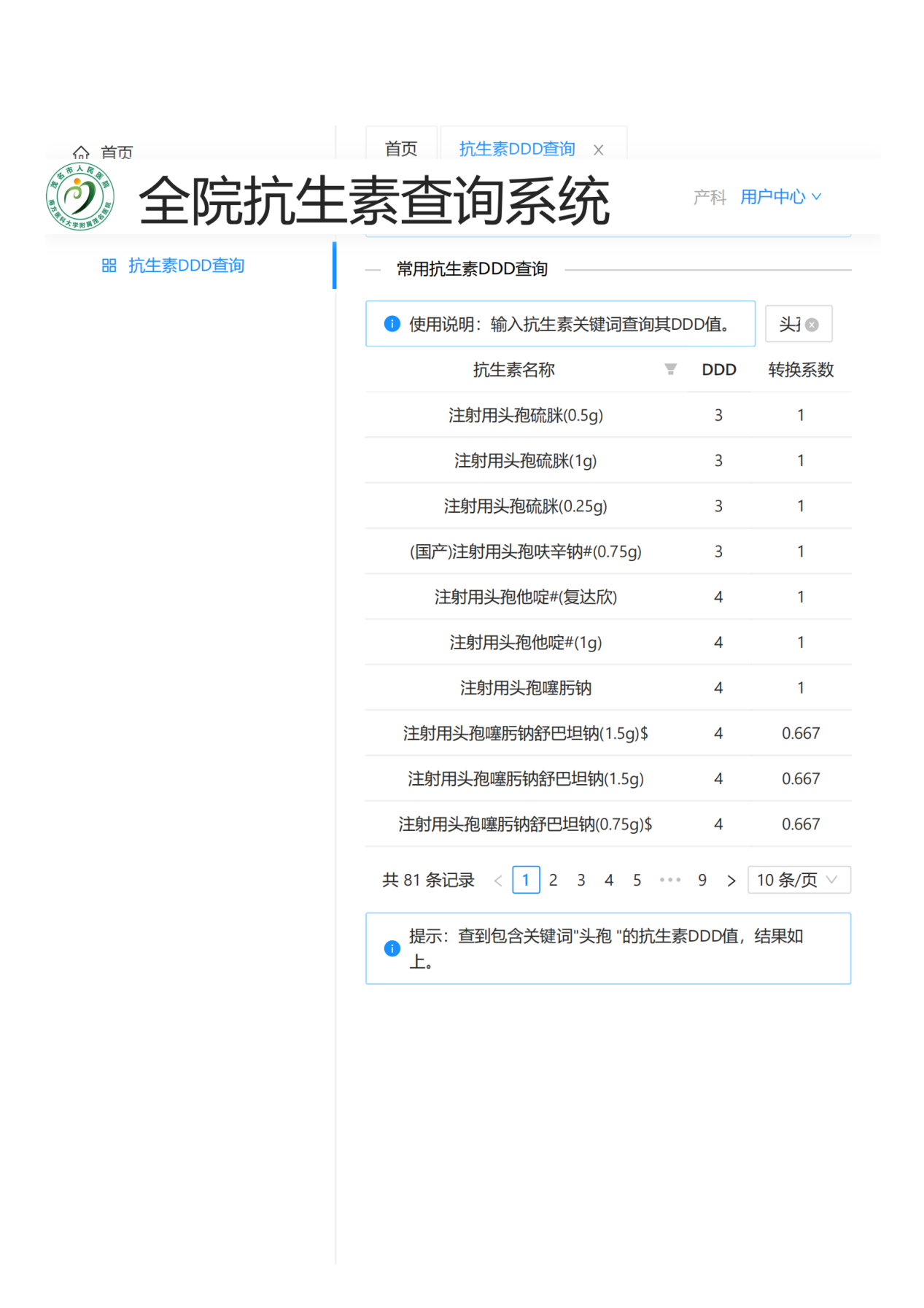
该系统经历了核心功能版的1.0版,后为方便各科室管理设置账号、密码登录,并美化了界面((图 18,图 19,图 20)。
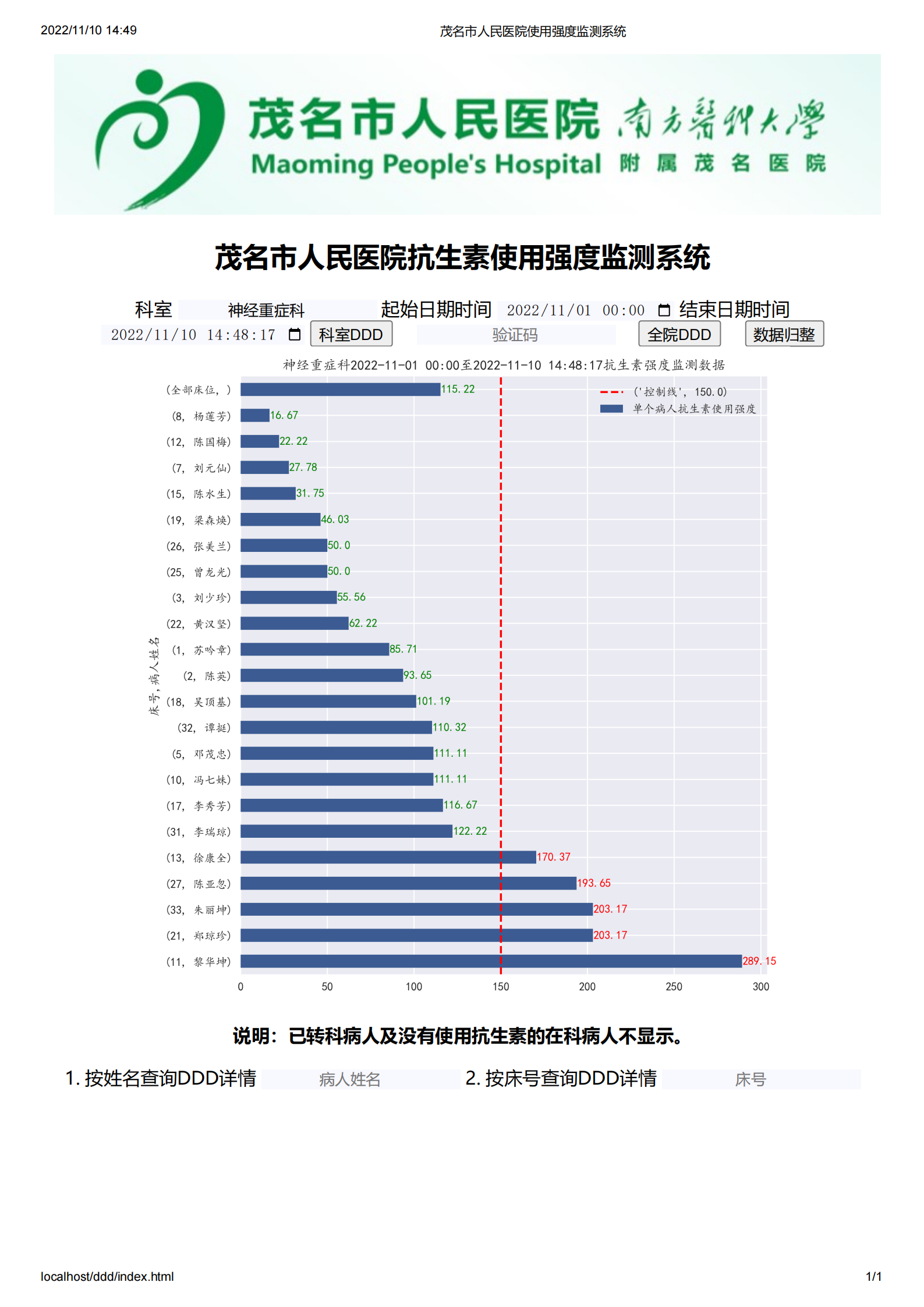
3.3 使用效果图
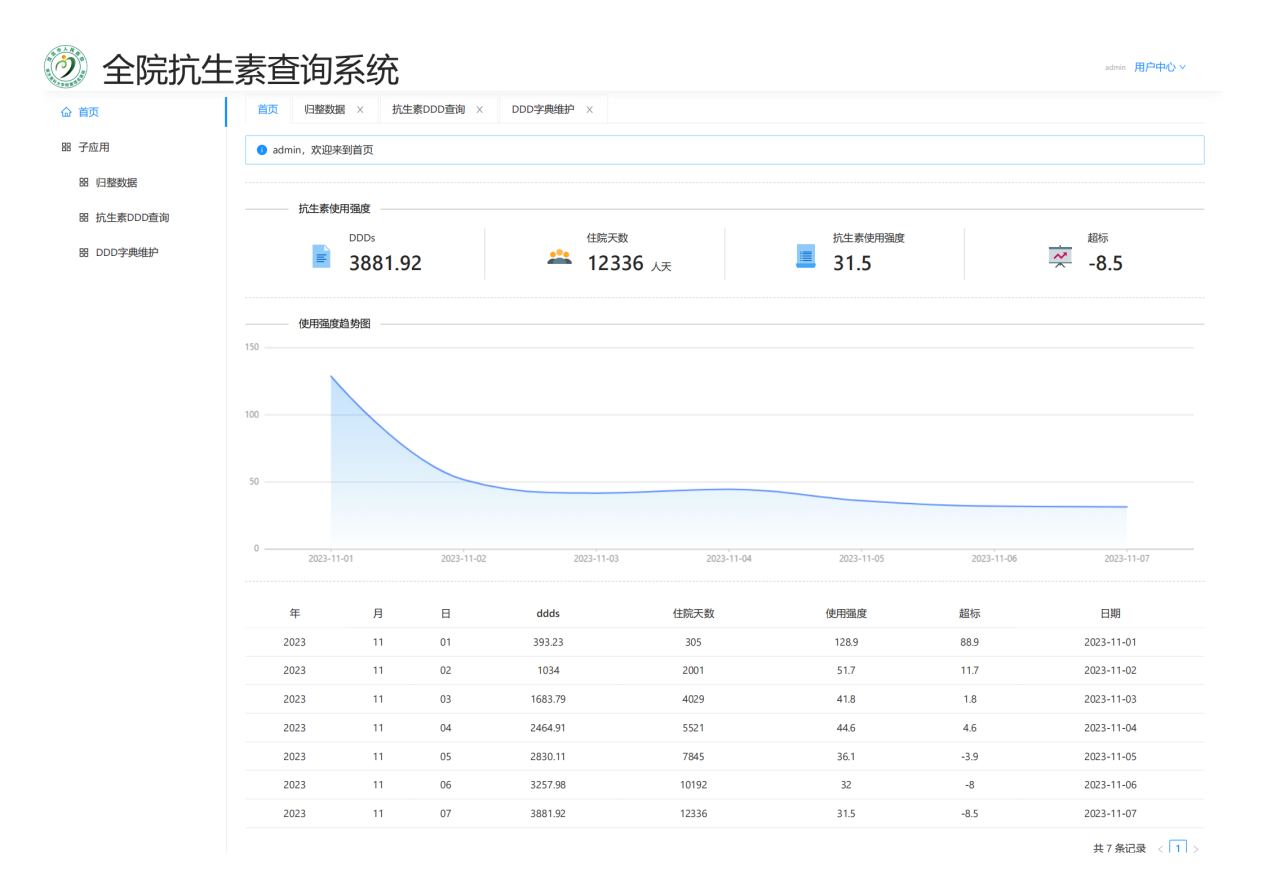
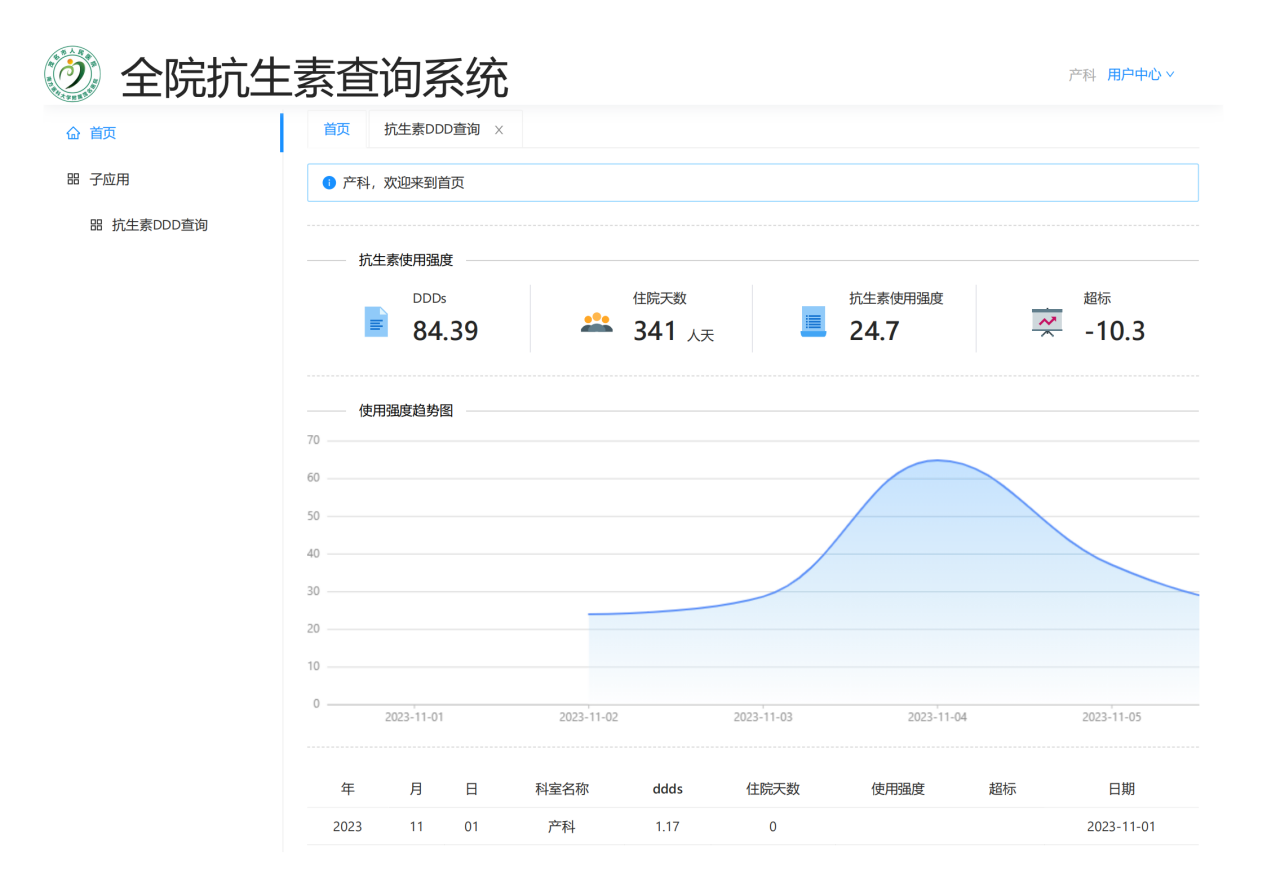
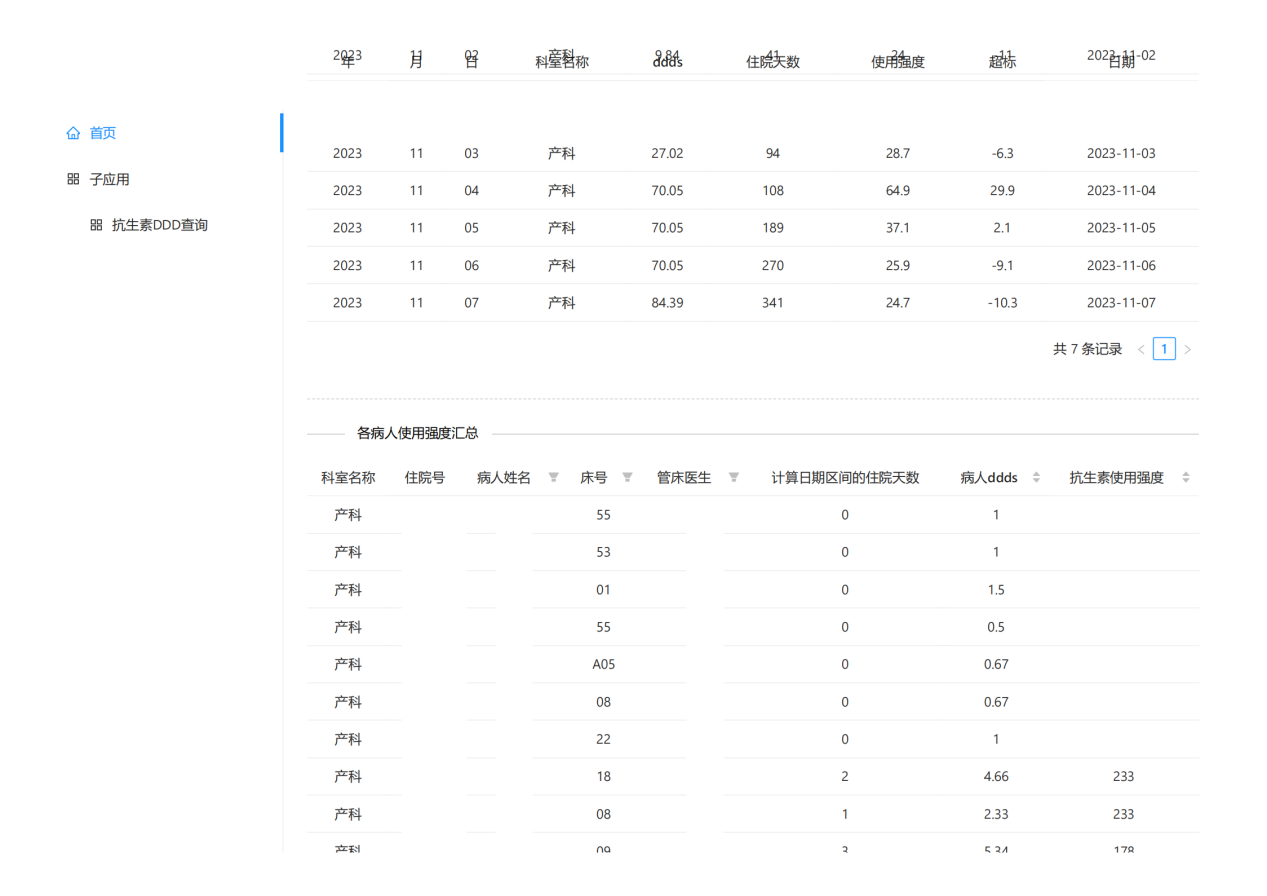
系统建成后极大方便医院管理层和临床医生对全院、科室、医生3个层次的抗生素使用强度的数据实时趋势的监测,随时按病情需要和监管需要调整抗生素的使用(图 21,图 22,图 23,图 24,图 25,图 26,图 27)。


3.4 系统逻辑架构
该系统的逻辑架构经过反复讨论和调试、试用,最终成型(图 28)。
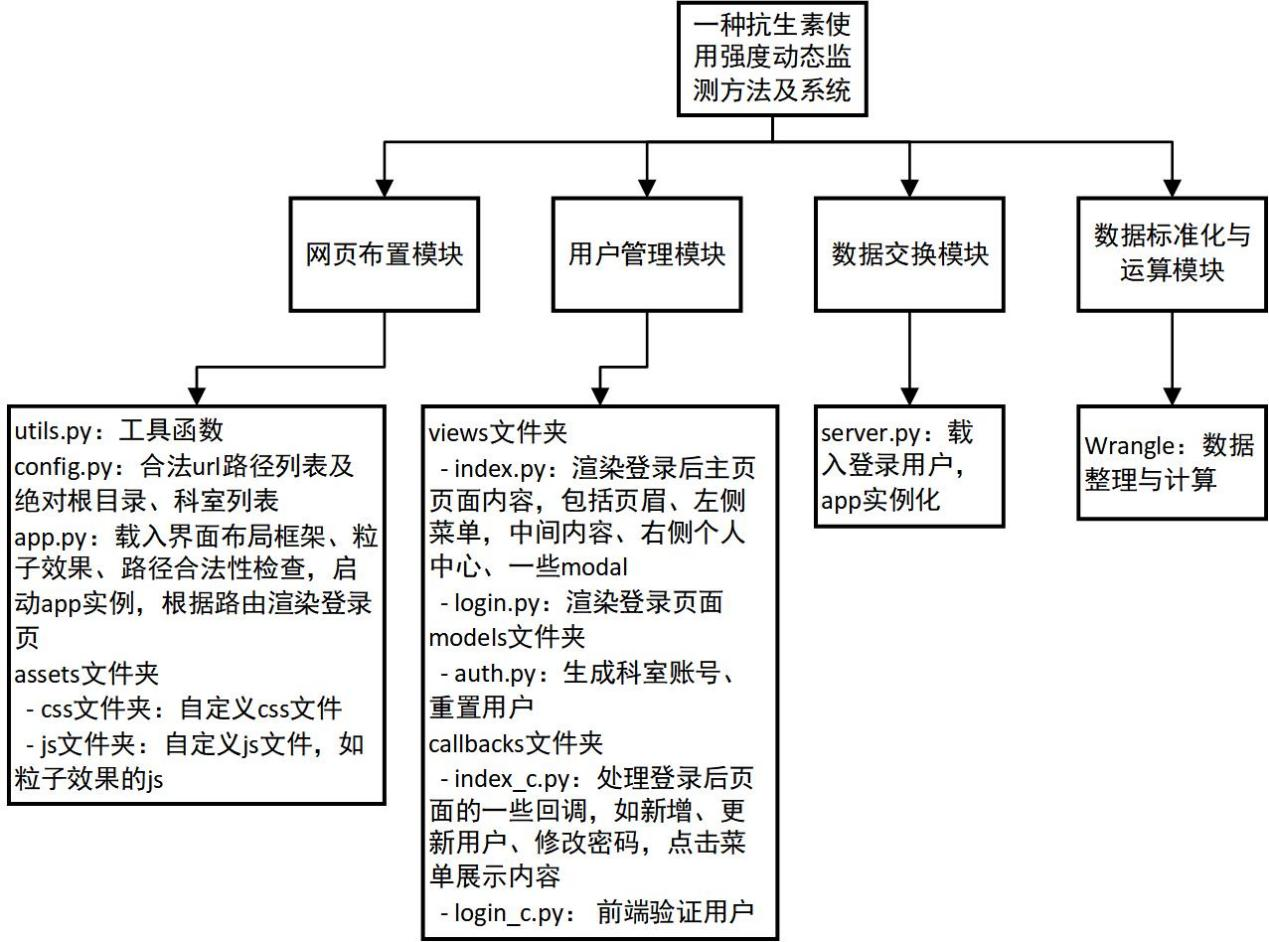
4 附件
4.1 申请与合同
- 专利申请(一种抗生素使用强度动态监测方法及系统).pdf
- 专利申请服务协议.pdf
4.2 代码
4.2.1 app.py
import dash, os
from dash import html, dcc
from dash.dependencies import Input, Output
from flask_login import current_user
import feffery_utils_components as fuc
from server import app
from config import RouterConfig
from flask import request
# 载入子页面
import views
app.layout = html.Div(
[
# 注入url监听,监听浏览器的地址的pathname
dcc.Location(id='url'),
fuc.FefferyExecuteJs(id='execute-js'),
# 背景粒子动画挂载点
html.Div(
id='particles-mount',
style={
'position': 'fixed',
'top': 0,
'left': 0,
'right': 0,
'bottom': 0,
'zIndex': -1
}
),
# 注入页面内容挂载点
html.Div(id='app-mount'),
# 路由重定向
html.Div(id='router-redirect-container'),
# 注入消息提示容器
html.Div(
[
# "用户管理-新增用户"消息提示
html.Div(id='index-user-manage-add-user-message-container'),
]
)
]
)
# 粒子效果
@app.callback(
Output('execute-js', 'jsString'),
Input('url', 'pathname')
)
def render_js(pathname):
print(pathname)
if pathname in ["/login", "/relogin"]:
return '''
// 第一个参数对应粒子效果挂载的容器id
particlesJS('particles-mount',
{
"particles": {
"number": {
"value": 80,
"density": {
"enable": true,
"value_area": 800
}
},
"color": {
"value": "#c7d2dd"
},
"shape": {
"type": "circle",
"stroke": {
"width": 0,
"color": "#000000"
},
"polygon": {
"nb_sides": 5
}
},
"opacity": {
"value": 0.5,
"random": false,
"anim": {
"enable": false,
"speed": 1,
"opacity_min": 0.1,
"sync": false
}
},
"size": {
"value": 5,
"random": true,
"anim": {
"enable": false,
"speed": 40,
"size_min": 0.1,
"sync": false
}
},
"line_linked": {
"enable": true,
"distance": 150,
"color": "#c7d2dd",
"opacity": 0.4,
"width": 1
},
"move": {
"enable": true,
"speed": 2,
"direction": "none",
"random": false,
"straight": false,
"out_mode": "out",
"attract": {
"enable": false,
"rotateX": 600,
"rotateY": 1200
}
}
},
"retina_detect": true,
"config_demo": {
"hide_card": false,
"background_color": "#b61924",
"background_image": "",
"background_position": "50% 50%",
"background_repeat": "no-repeat",
"background_size": "cover"
}
}
);
'''
else:
return dash.no_update
# 根据路由,生成不同的页面内容
@app.callback(
Output('app-mount', 'children'), # 页面内容
Output('router-redirect-container', 'children'), # 页面重定向,不清楚是什么
Input('url', 'pathname')
)
def router(pathname):
# 检验pathname合法性
if pathname not in RouterConfig.VALID_PATHNAME:
# 渲染404状态页
return [
views._404.render_content(),
None
]
# 检查当前会话是否已经登录
# 若已登录
if current_user.is_authenticated:
# 根据pathname控制渲染行为
if pathname == '/login':
# 重定向到主页面
return [
dash.no_update,
dcc.Location(
pathname='/',
id='router-redirect'
)
]
# 否则正常渲染主页面
return [
views.index.render_content(
current_user.id,
# current_user.gender,
current_user.role,
current_user.register_time
),
None
]
# 若未登录
# 根据pathname控制渲染行为
if pathname == '/login':
return [
views.login.render_content(),
None
]
elif pathname == '/relogin':
return [
views.login.render_content(relogin=True),
None
]
# 否则重定向到登录页
return [
dash.no_update,
dcc.Location(
pathname='/login',
id='router-redirect'
)
]
if __name__ == '__main__':
app.run(debug=True, port='8089')
4.2.2 server.py
import dash, os, shutil
from uuid import uuid4
from flask_login import LoginManager, UserMixin
from models.auth import UserAccount
from flask import request
app = dash.Dash(
__name__,
suppress_callback_exceptions=True,
update_title=None,
)
server = app.server
app.title = '茂名市人民医院抗生素使用强度查询监测系统'
app.favicon = 'favicon.ico'
# 初始化登陆管理类
login_manager = LoginManager()
login_manager.login_view = '/'
# 配置密钥
app.server.secret_key = 'ddd'
# 绑定纳入鉴权范围的flask实例
login_manager.init_app(server)
class User(UserMixin):
pass
@login_manager.user_loader
def load_user(user_id):
match_user = list(
UserAccount
.select()
.where(UserAccount.username == user_id)
.dicts()
)[0]
curr_user = User()
curr_user.id = user_id
curr_user.role = match_user['role']
# curr_user.gender = match_user['gender']
curr_user.register_time = match_user['register_time']
return curr_user
# 上传文件
@app.server.route('/upload/', methods=['POST'])
def upload():
'''
构建文件上传服务
:return:
'''
# 获取上传id参数,用于指向保存路径
uploadId = request.values.get('uploadId')
# 获取上传的文件名称
filename = request.files['file'].filename
# 流式写出文件到指定目录
filePath = os.path.join('data\dailyAntibioMedicalOrders', filename)
if os.path.exists(filePath): # 如果存在的话则备份,防止被覆盖
src_path = filePath
dst_path = f'data\\backup\\{uuid4()}-{filename}'
shutil.copy(src_path, dst_path)
with open(filePath, 'wb') as f:
# 流式写出大型文件,这里的10代表10MB
for chunk in iter(lambda: request.files['file'].read(1024 * 1024 * 10), b''):
f.write(chunk)
# print(imgFilePath)
return filePath4.2.3 config.py
import os
import pandas as pd
from utils import getFolderExcelFileInfo
import feffery_antd_components as fac
import feffery_utils_components as fuc
from datetime import datetime
from dash import html
class PathConfig:
# 项目绝对根目录
ABS_ROOT_PATH = os.path.abspath(os.getcwd())
class RouterConfig:
# 合法pathname列表
VALID_PATHNAME = [
'/', '/login', '/relogin'
]
class Departments:
deptlst = ['中医肛肠科', '中医针灸', '乳腺科', '产科', '儿科', '全科医学科', '关节四肢骨病外科', '内分泌科', '创伤骨科',
'口腔科', '呼吸与危重症一区', '呼吸与危重症二区', '外科ICU', '妇科一区', '妇科二区', '小儿外、疝外科',
'康复科', '心脏重症监护病房(CCU)', '心血管内一科', '心血管内二科', '心血管外科', '急诊ICU',
'急诊综合病区', '感染科', '手足显微外科', '新生儿科', '水东湾分院应急感染病一区', '泌尿外一科',
'泌尿外二科', '消化内一科', '消化内二科', '烧伤整形', '甲状腺血管外科', '皮肤科', '眼科', '神经内三科',
'神经内二科', '神经内科一区', '神经外一科', '神经外二科', '神经重症科', '综合科', '综合科特需病房',
'耳鼻喉科', '肝胆胰肛肠外科', '肾内二科', '肾内科', '肿瘤一科', '肿瘤二科', '肿瘤科三区',
'胃肠外科一区', '胃肠外科二区', '胸外科', '脊柱外一区', '脊柱外二区', '血液风湿科', '重症医学科',
'重症医学科三区','admin']
class Menu:
submenuItemsHosp = [ {
'component': 'Item',
'props': {
'key': '归整数据',
'title': '归整数据',
'icon': 'antd-app-store'
}
},
{
'component': 'Item',
'props': {
'key': '抗生素DDD查询',
'title': '抗生素DDD查询',
'icon': 'antd-app-store'
}
},
{
'component': 'Item',
'props': {
'key': 'DDD字典维护',
'title': 'DDD字典维护',
'icon': 'antd-app-store'
}
}
]
submenuItemsDept = [
{
'component': 'Item',
'props': {
'key': '抗生素DDD查询',
'title': '抗生素DDD查询',
'icon': 'antd-app-store'
}
},
]
class Table:
moFileInfo = getFolderExcelFileInfo('data/dailyAntibioMedicalOrders').sort_values(by = '文件', ascending=False)
moFileColumns, moFileData = [{'title': col,'dataIndex': col} for col in moFileInfo.columns], moFileInfo.to_dict(orient='records')
dddDict = pd.read_csv('dict/ddd测算字典.csv', dtype={'DDD':str, '转换系数':str})
filterOptionsHospDetail = {
'科室名称': {
'filterSearch': True
}
}
filterOptionsDeptDetail = {col: {
'filterSearch': True
} for col in ['病人姓名', '管床医生','抗生素名称', '医嘱类型','床号']
}
sortOptionsHospDetail = {
'sortDataIndexes': ['超标','使用强度']
}
sortOptionsDeptDetail = {
'sortDataIndexes': ['抗生素使用强度','病人ddds','DDD', '医嘱日期', '开抗生素时间', '停抗生素时间']
}
filterOptionsHospSummary = {
'科室名称': {
'filterSearch': True
}
}
filterOptionsDeptSummary = {col: {
'filterSearch': True
} for col in ['病人姓名', '管床医生','抗生素名称', '医嘱类型','床号']
}
sortOptionsHospSummary = {
'sortDataIndexes': ['超标','使用强度']
}
sortOptionsDeptSummary = {
'sortDataIndexes': ['抗生素使用强度','病人ddds','DDD']
}
class TabItems:
tabItemChildren = {}
tabItemChildrenDataNormalization = [ # 左对齐
fac.AntdDivider(
isDashed=True
),
fac.AntdCollapse(
[
# 左对齐
fac.AntdDivider(
'上传医嘱数据',
innerTextOrientation='left'
),
fac.AntdSpace(
[
fac.AntdAlert(
message='说明:选定要上传医嘱的日期后再上传。',
showIcon=True,
type='warning',
),
fac.AntdDatePicker(
id = 'moDateTime',
picker='date',
placeholder='选择医嘱日期',
# showTime=True,
style={
# 使用css样式固定宽度
'width': '10vw'
}
),
fac.AntdUpload(
id = 'uploadExcel',
apiUrl='/upload/',
fileMaxSize=10,
# multiple=True,
disabled=True,
fileTypes=['xlsx'],
buttonContent='上传数据'
),
]
),
fac.AntdDivider(
'已上传医嘱数据列表',
innerTextOrientation='left'
),
fac.AntdTable(
columns= Table.moFileColumns,
data = Table.moFileData,
id = 'uploadedExcelFileListTable',
# filterOptions = {
# '文件': {
# 'filterSearch': True
# }
# },
sortOptions={
'sortDataIndexes': ['文件']
},
)
],
title='展开/折叠上传医嘱数据',
isOpen=True,
ghost=False
),
fac.AntdDivider(
'归整医嘱数据',
innerTextOrientation='left'
),
fac.AntdSpace(
[
fac.AntdAlert(
message='说明:选定要归整医嘱日期后再归整。补归整时请先从最早日期开始归整。',
showIcon=True,
type='warning',
),
fac.AntdDatePicker(
id='normalizedDate',
picker = 'date',
# defaultValue = [datetime.today().strftime("%Y-%m-%d")] * 2 ,
placeholder='选择归整日期',
style={
# 使用css样式固定宽度
'width': '10.6vw'
}
),
fac.AntdButton(
[
fac.AntdIcon(
icon='fc-add-database'
),
'开始归整数据'
],
id = 'startNormalize'
),
]
),
fac.AntdSpin(
[
html.Div(
id='countUpDiv',
style = {'height':'100px'}
),
],
indicator=fuc.FefferyExtraSpinner(
type='wave'
),
text = '正在归整数据,稍等。'
),
html.Div(
id='normalizationMessageDiv',
style = {'height':'100px'}
),
]
tabItemChildrenDataQueryDDD = [ # 左对齐
fac.AntdDivider(
'常用抗生素DDD查询',
innerTextOrientation='left'
),
fac.AntdSpace(
[
fac.AntdAlert(
message='使用说明:输入抗生素关键词查询其DDD值。',
showIcon=True,
type='info',
),
fac.AntdInput(id = 'queryAntibioticName',
placeholder='输入抗生素名称',
allowClear=True,
style={
'width': '8vw',
}
)
]
),
fac.AntdTable(
columns= [{'title': col,'dataIndex': col} for col in ['抗生素名称','DDD','转换系数']],
data = [],
id = 'dddQueryResult',
filterOptions = {
'抗生素名称': {
'filterSearch': True
}
},
# sortOptions={
# 'sortDataIndexes': ['文件']
# },
),
html.Div(
id='normalizationMessageDiv',
style = {'height':'100px'}
),
]
tabItemChildren['归整数据'] = tabItemChildrenDataNormalization
tabItemChildren['抗生素DDD查询'] = tabItemChildrenDataQueryDDD4.2.4 utils.py
import hashlib
import os
import pandas as pd
from datetime import datetime
def str2md5(input_str: str):
md5 = hashlib.md5()
md5.update(input_str.encode('utf-8'))
return md5.hexdigest()
def getFolderExcelFileInfo(folder_path):
# 获取文件夹下所有文件的路径
files = [os.path.join(folder_path, f) for f in os.listdir(folder_path)]
# 遍历文件
file_info = []
for file_path in files:
# 过滤非 Excel 文件
if not file_path.endswith('.xlsx') and not file_path.endswith('.xls'):
continue
# 获取修改时间和文件大小
mtime = os.path.getmtime(file_path)
mtime = pd.to_datetime(mtime, unit='s').tz_localize('UTC').tz_convert('Asia/Shanghai').strftime('%Y-%m-%d %H:%M:%S') # 转换为当地时间
size = os.path.getsize(file_path)
size = f'{(size / 1024):.2f} KB'
# 将文件信息存储到 list 中
file_info.append({
'文件': os.path.basename(file_path),
'上传时间': mtime,
'大小': size
})
# 将文件信息构造成 DataFrame
df_file = pd.DataFrame(file_info)
# 输出 DataFrame
return df_file
def getFirstDayofCurrentMonth():
# 获取当前年月
now = datetime.now()
year_month = now.strftime('%Y-%m')
# 获取当月第一天
first_day_of_month = datetime.strptime(f'{year_month}-01', '%Y-%m-%d')
# 格式化为 "YYYY-MM-DD" 的字符串
formatted_date = first_day_of_month.strftime('%Y-%m-%d')
return formatted_date4.2.5 wrangle.py
import pandas as pd, numpy as np
import os, re
from datetime import datetime, timedelta
# 将医嘱频次转换为整数
def freq(freq:str):
if freq in ["Qd", "Qn", "St", "ST","Prn","特殊间隔", "S1"]:
freqN = 1
elif freq in ['Q12h', 'Bid','S2']:
freqN = 2
elif freq in ['Q8h', 'Tid']:
freqN = 3
elif freq in ['Q6h', 'Qid']:
freqN = 4
elif freq in ['Q4h']:
freqN = 6
elif freq in ['Qod']: # 两天1次
freqN = 0.5
elif freq in ['Q3d']: # 两天1次
freqN = 0.33
else:
freqN = np.nan
print(f"\r注意,产生频次为空的变量:{freq}", end='')
# 后期将警告存入日志文件,显示在前端再维护
return freqN
# 统一与合并数据
def unifyMergeData(start_date, end_date, year, month):
# start_date = '2023-04-24'
# end_date = '2023-04-24'
date_range = pd.date_range(start=start_date, end=end_date)
# 重整数据,由于去重的存在,重整数据不会导致重复计算
for date in date_range:
day = pd.Timestamp(date).date() #取日期部分,不要时间部分
xlsx = f'data/dailyAntibioMedicalOrders/抗生素医嘱-{day}.xlsx'
# 指定需要转换为字符串类型的列,统一参与去重的字段,才能有效去重
if not os.path.exists(xlsx): # 文件不存在,则跳过
print(f'{xlsx}不存在')
date_range = date_range.drop(date)
# print(date_range)
if not date_range.empty:
for date in date_range:
str_cols1 = ['科室名称', '住院号', '病人姓名', '床号', '管床医生', '医嘱类型', '抗生素医嘱', '抗生素名称',
'用量', '单位', '频次', '开抗生素时间', '停抗生素时间','转科信息','医嘱日期']
str_cols2 = ['科室名称', '住院号', '病人姓名', '床号', '管床医生', '医嘱类型', '抗生素医嘱', '抗生素名称',
'用量', '单位', '频次', '开抗生素时间', '停抗生素时间','转科信息','备注','医嘱日期']
day = pd.Timestamp(date).date() #取日期部分,不要时间部分
xlsx = f'data/dailyAntibioMedicalOrders/抗生素医嘱-{day}.xlsx'
# 读取 Excel 文件,并将指定列的数据类型全部转换为字符型
df = pd.read_excel(xlsx, dtype={col: str for col in str_cols1})
hosp = df[['科室名称', '住院号', '病人姓名', '床号', '管床医生', '医嘱类型', '抗生素名称', '抗生素医嘱',
'用量','单位','频次','开抗生素时间','停抗生素时间','转科信息']].copy()
hosp['备注'] = df.抗生素医嘱.apply(lambda x: '交长嘱使用' if str(x).__contains__('长嘱') else '').astype(str) # 交长嘱使用的医嘱,不应计入,作出标记
hosp['医嘱日期'] = str(day) # 用来计算床天数,
# 但有转科,则开医嘱的时间跟医嘱日期不同,导致少算1天,通过'住院号', '医嘱类型','抗生素名称','用量','单位','频次','开抗生素时间'可匹配到转入科室和转出科室的医嘱所在的日期,
# 即可反映实际住院时间的医嘱日期
# 判断该月份抗生素归整数据是否存在,不存在则创建
if not os.path.exists(f'data/wrangledAntibioMedcalOrders/全院/抗生素使用医嘱汇总-{year}年{month}月.csv'):
# 获取当前工作目录
current_path = os.getcwd()
# 定义目标文件夹路径
target_path = os.path.join(current_path, 'data/wrangledAntibioMedcalOrders/全院/')
# 创建文件夹
os.makedirs(target_path, exist_ok=True)
updatedCSV = hosp
# 统一参与去重的字段,才能有效去重
updatedCSV.开抗生素时间 = updatedCSV.开抗生素时间.apply(lambda x:pd.Timestamp(x).strftime('%Y-%m-%d %H:%M:%S.%f')[:-3] if not pd.isna(x) else str(x).strip())
# 顺便统一停抗生素时间
updatedCSV.停抗生素时间 = updatedCSV.停抗生素时间.apply(lambda x:pd.Timestamp(x).strftime('%Y-%m-%d %H:%M:%S.%f')[:-3] if not pd.isna(x) else str(x).strip())
# 空值的处理和日期时间格式的统一是彻底去重的关键
updatedCSV.fillna('',inplace=True)
updatedCSV.停抗生素时间 = updatedCSV.停抗生素时间.str.replace('nan','')
updatedCSV.开抗生素时间 = updatedCSV.开抗生素时间.str.replace('nan','')
# updatedCSV.医嘱日期 = updatedCSV.医嘱日期.astype(str)
updatedCSV.drop_duplicates(subset=str_cols1, keep='first', inplace=True)
updatedCSV.to_csv(f'data/wrangledAntibioMedcalOrders/全院/抗生素使用医嘱汇总-{year}年{month}月.csv', index=False)
else: #追加至已有抗生素医嘱汇总表,并去重
# 读取CSV文件,并将指定列的数据类型全部转换为字符型
existedCSV = pd.read_csv(f'data/wrangledAntibioMedcalOrders/全院/抗生素使用医嘱汇总-{year}年{month}月.csv',dtype={col: str for col in str_cols2})
# # 新旧纵向合并
# updatedCSV = pd.concat([existedCSV, hosp])
# 新旧纵向合并
updatedCSV = pd.merge(existedCSV, hosp, on=str_cols2, how='outer') # on全部列
# 统一参与去重的字段,才能有效去重
updatedCSV.开抗生素时间 = updatedCSV.开抗生素时间.apply(lambda x:pd.Timestamp(x).strftime('%Y-%m-%d %H:%M:%S.%f')[:-3] if not pd.isna(x) else str(x).strip())
# 顺便统一停抗生素时间
updatedCSV.停抗生素时间 = updatedCSV.停抗生素时间.apply(lambda x:pd.Timestamp(x).strftime('%Y-%m-%d %H:%M:%S.%f')[:-3] if not pd.isna(x) else str(x).strip())
# 空值的处理和日期时间格式的统一是彻底去重的关键
updatedCSV.fillna('',inplace=True)
updatedCSV.停抗生素时间 = updatedCSV.停抗生素时间.str.replace('nan','')
updatedCSV.开抗生素时间 = updatedCSV.开抗生素时间.str.replace('nan','')
# print(updatedCSV.columns)
updatedCSV.医嘱日期 = updatedCSV.医嘱日期.astype(str)
# 更新,需要去重,但要是全部字段相同的才去重,前提是保证字段的统一性以保证去重的可靠性
updatedCSV.drop_duplicates(subset=str_cols1, keep='first', inplace=True) # keep=first或last均不影响
# updatedCSV = updatedCSV.sort_values(by=['科室名称', '住院号','医嘱类型', '抗生素名称'])
updatedCSV.to_csv(f'data/wrangledAntibioMedcalOrders/全院/抗生素使用医嘱汇总-{year}年{month}月.csv', index=False)
return updatedCSV
else:
return pd.DataFrame()
# 根据转科信息生成住科区间数据
def generateDeptStayRange(patID, transInfo):
# transInfo = '从神经重症科 于 2023年03月23日16时45分 转往 神经外二科 神经外科二区,从神经外二科 于 2023年03月25日20时43分 转往 神经重症科 神经重症医学科,从神经重症科 于 2023年03月31日16时00分 转往 呼吸与危重症一区 呼吸与危重症一区'
# 定义正则表达式模式
pattern_start = r'从(.*?)\s于'
pattern_end = r'转往\s(.*?)\s'
# 匹配出所有符合要求的科室名称
start_depts = re.findall(pattern_start, transInfo)
end_depts = re.findall(pattern_end, transInfo)
# 输出结果
lst = list(zip(start_depts, end_depts))
dept_list = [item for pair in lst for item in pair]
# 匹配日期时间
pattern_datetime = r'\d{4}年\d{2}月\d{2}日\d{2}时\d{2}分'
lst = re.findall(pattern_datetime, transInfo)
dt_list = [i for i in lst for _ in range(2)]
# 科室与转科时间
dept_dt = pd.DataFrame(zip(dept_list, dt_list), columns=['科室名称','转科时间'])
# # 比较相邻两行科室名称是否相同
mask = dept_dt['科室名称'].ne(dept_dt['科室名称'].shift())
# # 根据相邻相同科室名称行分组的标签
label = mask.cumsum()
# # 将同一科室名称下的转科时间用"~"进行拼接
dept_dt = dept_dt.groupby(label).agg({'科室名称': 'first', '转科时间': lambda x: '~'.join(x)}).rename(columns={'转科时间': '住科期间'})
dept_dt.iloc[0,1] = "~" + dept_dt.iloc[0,1] # 第一行为转出时间
dept_dt.iloc[-1,1] = dept_dt.iloc[-1,1] + "~" # 最后一行为转入时间
dept_dt.reset_index(drop=True, inplace=True)
# 将相同科室名称的转科时间通过"~"连接
dept_dt = dept_dt.groupby('科室名称')['住科期间'].apply(lambda x: ','.join(x)).reset_index()
dept_dt['住院号'] = patID
return dept_dt
# 根据停止计算床天数日期时间调整住科期间,适用于科室调整关闭的情况
def calStoppedDateTimeIntervals(stay_time, stop_datetime):
# # 日期时间区间
# stay_time = '2023-04-11~2023-04-17 00:06,2023-04-18 16:00~2023-04-21 10:54'
# # 截止日期时间
# stop_datetime = '2023-04-20 08:00'
dateTimeIntervals = stay_time.split(',')
stoppedDateTimeIntervals = []
for interval in dateTimeIntervals:
start_time, end_time = interval.split('~')
if pd.to_datetime(end_time) > pd.to_datetime(stop_datetime) > pd.to_datetime(start_time): # 如果落在时间区间内才有效
interval = '~'.join([stop_datetime, end_time]) # 用stop_datetime替换start_time,起到截断作用
stoppedDateTimeIntervals.append(interval)
return ','.join(stoppedDateTimeIntervals)
#由转科信息提取住科期间数据
def extractDeptStayRangeInfo(updatedCSV):
# 以住院号分组,找出最完整转科信息的行
transLongestInfo = (
updatedCSV
.query('转科信息 != ""') # 排除空字符行
.groupby(['住院号'])['转科信息'] #按住院号分组,对转科信息进行聚合计算
.agg(lambda x: max(x, key=len)) # 求字符串最长的行
.reset_index() # 将Series转换为Dataframe
)
# 提取转科信息,得到住科期间数据,供匹配
DeptStayRangeList = []
for patID, transInfo in transLongestInfo.values:
# print(patID, transInfo)
DeptStayRange = generateDeptStayRange(patID, transInfo)
DeptStayRangeList.append(DeptStayRange)
DeptStayRangeInfo = pd.concat(DeptStayRangeList, ignore_index=True)
if DeptStayRangeInfo.duplicated().any():
print('有重复行')
else:
print('无重复行,科室和住院号分组,住科期间信息是唯一且完整的。')
DeptStayRangeInfo.住科期间.replace({'[年月]':'-', '日':' ', '时':':', '分':''}, regex=True, inplace=True) # 转换格式
#与停止计算日期时间比对,得到更新后的住科期间数据
# 增加1列“不计算起始日期”
stopCalDeptDate = pd.read_csv('dict/不计算床天科室名称及日期.csv')
DeptStayRangeInfo = DeptStayRangeInfo.merge(stopCalDeptDate, how='left')
DeptStayRangeInfo.loc[DeptStayRangeInfo.不计算起始日期.notna(), '住科期间'] = (DeptStayRangeInfo.loc[DeptStayRangeInfo.不计算起始日期.notna(), ['住科期间', '不计算起始日期']]
.apply(lambda row:calStoppedDateTimeIntervals(row['住科期间'], row['不计算起始日期']), axis=1)
)
return DeptStayRangeInfo
# 计算住院天数的函数2,普适性好
def calcStayDays2(stayRangeInfo:str, startDate, endDate):
# start_date, end_date分别是医嘱日期的最小值和最大值
# 原始字符串
# stayRangeInfo = '2023-04-11~2023-04-17 00:06,2023-04-18 16:00~2023-04-21 10:54'
# print('stayRangeInfo:', stayRangeInfo)
startDate = pd.to_datetime(startDate).date()
endDate = pd.to_datetime(endDate).date()
# 分割成不同的日期区间
date_ranges = stayRangeInfo.split(',')
# 处理每个日期区间
date_intervals = []
for date_range in date_ranges:
# 提取起始日期和结束日期
left_str, right_str = date_range.split('~')
# 转换成 date 类型
left_date = pd.to_datetime(left_str).date()
right_date = pd.to_datetime(right_str).date()
# 存储成 (left_date, right_date) 的形式
date_intervals.append((left_date, right_date))
# print(date_intervals)
# 初始值为 0
overlap_days = 0
# 计算与每个日期区间的重复天数
for interval in date_intervals:
# 如果两个日期区间没有交集,则直接跳过
if endDate < interval[0] or startDate > interval[1]:
continue
# 计算重复天数
overlap_start = max(startDate, interval[0])
overlap_end = min(endDate, interval[1])
overlap_days += (overlap_end - overlap_start).days
# 打印结果
# print('要计算的日期区间:', startDate, '至', endDate)
# print('不连续的日期区间:', date_intervals)
# print('与这些日期区间重复的天数为:', overlap_days)
if overlap_days == 0:
overlap_days = 1
return overlap_days
# 自定义函数,用于判断临嘱的开抗生素时间是否落在住科期间的时间区间内
def is_within_DeptStay_interval(row):
# 解析住科期间的时间区间
intervals = row['住科期间'].split(',')
for interval in intervals:
start_str, end_str = interval.split('~')
start_date = pd.to_datetime(start_str)
end_date = pd.to_datetime(end_str)
# 判断开抗生素时间是否在住科期间的时间区间内
if start_date <= row['开抗生素时间'] <= end_date and row['医嘱类型'] == '临嘱':
return True
return False
# 自定义函数,用于判断临嘱的开抗生素时间是否落在当月的时间区间内
def is_within_CurrentMonth_interval(row, year, month, last_day):
# 解析住科期间的时间区间
firstDay = pd.Timestamp(year=year, month=month, day=1, hour=0, minute=0, second=0)
lastDay = pd.Timestamp(year=year, month=month, day=last_day, hour=23, minute=59, second=59)
# 判断开抗生素时间是否在当月的时间区间内
if firstDay <= row['开抗生素时间'] <= lastDay and row['医嘱类型'] == '临嘱':
return True
return False
# 计算住科期间抗生素使用日期区间的函数
def calcDateTimeRange(start_ab, end_ab, stay_time):
# start_ab, end_ab分别为开、停抗生素时间,stay_time为住科期间
# 定义数据
# start_ab = '2023-04-12 03:44:14.575000'
# end_ab = '2023-04-12 08:00:12.548'
# stay_time = '2023-04-11~2023-04-17 00:06,2023-04-18 16:00~2023-04-21 10:54'
# 转化为datetime对象
start_ab_dt = pd.to_datetime(start_ab)
end_ab_dt = pd.to_datetime(end_ab)
# 分割住科期间为单独的时间段
stay_periods = stay_time.split(',')
stay_intervals = []
for period in stay_periods:
if '~' in period:
start, end = period.split('~')
start_dt = pd.to_datetime(start)
end_dt = pd.to_datetime(end)
if start_dt < end_dt: # 如果住科期间的入科时间(即计算区间的开始时间)迟于出科时间,则该住科期间肯定不在计算区间内,不需处理
stay_intervals.append(pd.Interval(start_dt, end_dt))
else:
# print(stay_time)
pass
else:
start_dt = pd.to_datetime(period)
stay_intervals.append(pd.Interval(start_dt, start_dt))
# 计算交集
CalcDateTimeRange = []
for interval in stay_intervals:
intersect_start = max(start_ab_dt, interval.left)
intersect_end = min(end_ab_dt, interval.right)
if intersect_start <= intersect_end:
intersection = pd.Interval(intersect_start, intersect_end)
CalcDateTimeRange.append("~".join([str(intersection.left), str(intersection.right)]))
# print('开抗生素时间至停抗生素时间与住科期间的交集:', intersection)
else:
# print('开抗生素时间至停抗生素时间与住科期间没有交集')
pass
return ','.join(CalcDateTimeRange)
def getCurrentMonthStayRange(interval_str, year, month, last_day):
intervals = interval_str.split(',')
overlapping_intervals = []
for interval in intervals:
start_str, end_str = interval.split('~')
start_date = pd.to_datetime(start_str)
end_date = pd.to_datetime(end_str)
# 判断区间是否与4月份重叠
if start_date <= pd.Timestamp(year=year, month=month, day=last_day) and end_date >= pd.Timestamp(year=year, month=month, day=1):
# 计算重叠时间区间
overlapping_start = max(start_date, pd.Timestamp(year=year, month=month, day=1))
overlapping_end = min(end_date, pd.Timestamp(year=year, month=month, day=last_day))
overlapping_interval = f"{overlapping_start}~{overlapping_end}"
overlapping_intervals.append(overlapping_interval)
return ','.join(overlapping_intervals)
# 按照当月抗生素使用时间区间计算抗生素使用的天数,若为空值则为0
def calCurrentMonthAntiBioticsUseDays(CurrentMonthAntiBioticsUseIntervals:str):
if CurrentMonthAntiBioticsUseIntervals:
totalDays = 0
for interval in CurrentMonthAntiBioticsUseIntervals.split(','):
duration = pd.to_datetime(interval.split('~')[1]) - pd.to_datetime(interval.split('~')[0])
days = duration.days + duration.seconds / (24 * 3600)
totalDays += days
if totalDays<0:
totalDays = 0
return round(totalDays, 3)
else:
# print(CurrentMonthAntiBioticsUseIntervals)
return 0
# 下面归整
def wrangleData(start_date, end_date, year, month, day):
# start_date = '2023-04-25'
# end_date = '2023-04-26'
# year, month = '2023', '04'
updatedCSV = unifyMergeData(start_date, end_date, year, month)
if updatedCSV.empty:
print('无新数据归整')
print(len(updatedCSV))
# 提取住科期间数据
# 匹配停止计算时间
stopCalDeptDate = pd.read_csv('dict/不计算床天科室名称及日期.csv')
updatedCSV = updatedCSV.merge(stopCalDeptDate, how='left')
# 住科期间信息
DeptStayRangeInfo = extractDeptStayRangeInfo(updatedCSV) # 考虑到了Stopdate
# 先处理长嘱抗生素停嘱时间
mask = updatedCSV.医嘱类型=='长嘱'
# fillna的坑
updatedCSV.loc[mask, '停抗生素时间'] = \
updatedCSV.loc[mask, '停抗生素时间'].replace('', np.nan)
# 将同一组的抗生素时间双向填充,得到实际的长嘱停嘱时间
updatedCSV.loc[mask, '停抗生素时间'] = (
updatedCSV[mask]
.groupby(['科室名称', '住院号', '抗生素名称', '开抗生素时间'])['停抗生素时间'] # 这里不能缺了科室名称
.apply(lambda x: x.fillna(method = 'ffill').fillna(method = 'bfill')) # 组内双向填充,不会漏
)
# 剩下为空值的停抗生素时间为未停的抗生素,统一设置为当天晚上的23:59:59 @ 导致当天出院,第2天导出医嘱没有该病人,导致长嘱停嘱时间为空,被填充为最新日期的情况
#@ 改为该病人医嘱日期最大值那天的23:59:59
# maxMedicalOrderDateTime = datetime.now().replace(hour=23, minute=59, second=59, microsecond=0)
maxMedicalOrderDateDF = updatedCSV[mask].groupby(['科室名称', '住院号'])['医嘱日期'].agg(lambda x : f'{max(x)} 23:59:59').reset_index().rename(columns={'医嘱日期':'最新医嘱日期'})
updatedCSV = updatedCSV.merge(maxMedicalOrderDateDF, how='left')
updatedCSV.loc[mask & updatedCSV['停抗生素时间'].isna(),'停抗生素时间'] = updatedCSV.loc[mask & updatedCSV['停抗生素时间'].isna(),'最新医嘱日期']
###### 住院天数情况
# 医嘱信息与住科期间信息拼接
updatedCSV2 = updatedCSV.merge(DeptStayRangeInfo, how='left')
# 住科信息非空与空分开处理,处理结果合并即可,这两类病人无重叠
updatedCSV2.replace('', np.nan, inplace=True) # 注意,深坑,
updatedCSV2_IS_StayRangeInfo, updatedCSV2_Not_StayRangeInfo = updatedCSV2.query('住科期间.notnull()').copy(), updatedCSV2.query('住科期间.isnull()').copy()
## 先处理住科信息非空(有转科的病人)的情况
# 补齐住科期间的开区间,医嘱日期最小值和最大值,分别补齐左侧和右侧开区间,这里根据需要可增加"时分秒"
# 按住院号分组进行操作,并使用 lambda 表达式
updatedCSV2_IS_StayRangeInfo = updatedCSV2_IS_StayRangeInfo.groupby('住院号').apply(lambda x: x.assign(
住科期间=x['住科期间'].apply(lambda y: x['医嘱日期'].min() + y if y.startswith('~') else y).apply(lambda y: y + x['医嘱日期'].max() if y.endswith('~') else y)
))
# 按住院号分组,生成计算日期区间和计算日期区间的住院天数
# 按住院号分组进行操作,并使用 lambda 表达式
updatedCSV2_IS_StayRangeInfo = updatedCSV2_IS_StayRangeInfo.groupby('住院号').apply(lambda x: x.assign(
计算日期区间=f"{x['医嘱日期'].min()}~{x['医嘱日期'].max()}",
计算日期区间的住院天数=x['住科期间'].apply(calcStayDays2, startDate=x['医嘱日期'].min(), endDate=x['医嘱日期'].max())
))
##### 以上为住科信息非空的集合,计算床天数完成,
##### 下面计算住科信息为空的集合的床天数,
# # 非计算日期内的科室,要按不计算起始日期开始计算床天数,如日间化疗中心、血液净化中心等
updatedCSV2_Not_StayRangeInfo[['不计算起始日期', '医嘱日期']] = updatedCSV2_Not_StayRangeInfo[['不计算起始日期', '医嘱日期']].astype('datetime64[ns]')
updatedCSV2_Not_StayRangeInfo = updatedCSV2_Not_StayRangeInfo.groupby('住院号').apply(lambda x: x.assign(
计算日期区间的住院天数= (0 if x['不计算起始日期'].min() <= x['医嘱日期'].min() else
(x['医嘱日期'].max() - x['不计算起始日期'].min()).days if x['不计算起始日期'].min() <= x['医嘱日期'].max() else
(x['医嘱日期'].max() - x['医嘱日期'].min()).days)))
# updatedCSV2_Not_StayRangeInfo = updatedCSV2_Not_StayRangeInfo.groupby('住院号').apply(lambda x: x.assign(
# 计算日期区间的住院天数= (0 if pd.to_datetime(x['不计算起始日期'].min()) <= pd.to_datetime(x['医嘱日期'].min()) else
# (pd.to_datetime(x['医嘱日期'].max()) - pd.to_datetime(x['不计算起始日期'].min())).days if pd.to_datetime(x['不计算起始日期'].min()) <= pd.to_datetime(x['医嘱日期'].max()) else
# (pd.to_datetime(x['医嘱日期'].max()) - pd.to_datetime(x['医嘱日期'].min())).days)))
###### 处理有转科信息病人的抗生素使用情况,
# 分两种情况:有开抗生素医嘱时间和无开抗生素时间
# 先去重,减小计算量,不影响正确结果
updatedCSV2_IS_StayRangeInfo.drop_duplicates(subset=['科室名称', '住院号', '抗生素名称','开抗生素时间'], inplace=True)
# 将字符串类型的时间转换成 Timestamp 类型,后面有去重和计算要小心类型不一致
updatedCSV2_IS_StayRangeInfo['开抗生素时间'] = pd.to_datetime(updatedCSV2_IS_StayRangeInfo['开抗生素时间'])
updatedCSV2_IS_StayRangeInfo_IS_StartTime, updatedCSV2_IS_StayRangeInfo_Not_StartTime = updatedCSV2_IS_StayRangeInfo.query('开抗生素时间.notnull()').copy(), updatedCSV2_IS_StayRangeInfo.query('开抗生素时间.isnull()').copy()
# 处理有开抗生素时间的医嘱
# 判断抗生素归属科室是否正确。原因是有些抗生素是上一个科室或下一个科室的医嘱,却归在当前科室,开抗生素时间和停抗生素时间跟住科期间的关系来判断
# 处理临嘱,相对容易
# 应用自定义函数,这里不需要分组
# 判断临嘱是否是住科期间的
updatedCSV2_IS_StayRangeInfo_IS_StartTime.loc[updatedCSV2_IS_StayRangeInfo_IS_StartTime.医嘱类型=='临嘱', '是否住科期间内'] = \
updatedCSV2_IS_StayRangeInfo_IS_StartTime.apply(is_within_DeptStay_interval, axis=1)
# 判断临嘱是否是本月住科期间的
# 获取当前时间
# now = datetime.now()
# 获取当前月份的year和month
# year = now.year
# month = now.month
year = datetime.strptime(year, '%Y').year
month = datetime.strptime(month, '%m').month
# 获取当前月份的最后一天的day
last_day = (datetime(year, month+1, 1) - timedelta(days=1)).day
updatedCSV2_IS_StayRangeInfo_IS_StartTime.loc[updatedCSV2_IS_StayRangeInfo_IS_StartTime.医嘱类型=='临嘱', '是否当月住科期间内'] = \
updatedCSV2_IS_StayRangeInfo_IS_StartTime.apply(lambda row : is_within_CurrentMonth_interval(row, year=year, month=month, last_day=last_day), axis=1)
# 是否住科期间内跟是否当月住科期间内,同时为True时,为当月内有效医嘱(相对于当时住的科室是有效的)
updatedCSV2_IS_StayRangeInfo_IS_StartTime.loc[updatedCSV2_IS_StayRangeInfo_IS_StartTime.医嘱类型=='临嘱', '当月内有效医嘱'] = \
updatedCSV2_IS_StayRangeInfo_IS_StartTime.loc[updatedCSV2_IS_StayRangeInfo_IS_StartTime.医嘱类型=='临嘱',['是否住科期间内','是否当月住科期间内']]\
.apply(lambda row:all(row), axis=1)
#处理长嘱,相对复杂
### 注意,停抗生素时间的空字符不是空值,用fillna处理不了,因此先将''替换成np.nan,深坑二
mask = updatedCSV2_IS_StayRangeInfo_IS_StartTime.医嘱类型=='长嘱'
# updatedCSV2_IS_StayRangeInfo_IS_StartTime.loc[mask, '停抗生素时间'] = \
# updatedCSV2_IS_StayRangeInfo_IS_StartTime.loc[mask, '停抗生素时间'].replace('', np.nan)
# 将同一组的行打上相同的标签,标签作为新的一列加入 DataFrame
updatedCSV2_IS_StayRangeInfo_IS_StartTime.loc[mask, '长嘱组标签']= \
updatedCSV2_IS_StayRangeInfo_IS_StartTime[mask]\
.groupby(['住院号', '抗生素名称', '开抗生素时间']).ngroup().apply(lambda x: str(x))
# 计算开抗生素时间和停抗生素时间在住科期间的叠加区间
# 注意,会产生空字符,updatedCSV2_IS_StayRangeInfo_IS_StartTime['医嘱类型']=='长嘱'可以优化为mask,不用重复筛选
updatedCSV2_IS_StayRangeInfo_IS_StartTime.loc[
mask, '住科期间抗生素使用时间区间'] = \
updatedCSV2_IS_StayRangeInfo_IS_StartTime.loc[mask,['开抗生素时间', '停抗生素时间','住科期间']]\
.apply(lambda x: calcDateTimeRange(x['开抗生素时间'], x['停抗生素时间'], x['住科期间']), axis=1)
# updatedCSV2_IS_StayRangeInfo_IS_StartTime['住科期间抗生素使用时间区间'].replace('', np.nan, inplace=True)
# 计算长嘱的当月抗生素使用时间区间,#注意,会产生空字符(函数返回的)
updatedCSV2_IS_StayRangeInfo_IS_StartTime.loc[
(~updatedCSV2_IS_StayRangeInfo_IS_StartTime.住科期间抗生素使用时间区间.eq('')) & (mask),
'当月抗生素使用时间区间'] = \
updatedCSV2_IS_StayRangeInfo_IS_StartTime.loc[
(~updatedCSV2_IS_StayRangeInfo_IS_StartTime.住科期间抗生素使用时间区间.eq('')) & (mask),
'住科期间抗生素使用时间区间']\
.apply(lambda x: getCurrentMonthStayRange(x, year=year, month=month, last_day=last_day))
# # '当月抗生素使用时间区间'为空字符的,'是否当月住科期间内'为False,不为空字符则为True
updatedCSV2_IS_StayRangeInfo_IS_StartTime.loc[mask, '是否当月住科期间内'] = \
~updatedCSV2_IS_StayRangeInfo_IS_StartTime.loc[mask, '住科期间抗生素使用时间区间'].eq('')
updatedCSV2_IS_StayRangeInfo_IS_StartTime.loc[
mask, '当月内有效医嘱'] = updatedCSV2_IS_StayRangeInfo_IS_StartTime.loc[
mask, '是否当月住科期间内']
# 筛选有效住科期间的数据
updatedCSV2_IS_StayRangeInfo_IS_StartTime = updatedCSV2_IS_StayRangeInfo_IS_StartTime.query('当月内有效医嘱==True')
# # 计算抗生素使用强度
# 计算使用频次
updatedCSV2_IS_StayRangeInfo_IS_StartTime['频次_int'] = updatedCSV2_IS_StayRangeInfo_IS_StartTime.频次.apply(freq)
# 计算使用天数,临嘱为1天
updatedCSV2_IS_StayRangeInfo_IS_StartTime.loc[updatedCSV2_IS_StayRangeInfo_IS_StartTime['医嘱类型']=='临嘱', '使用天数'] = 1
# 计算使用天数,长嘱按“当月抗生素使用时间区间”计算,可能有多个区间。
updatedCSV2_IS_StayRangeInfo_IS_StartTime.loc[
mask, '使用天数'] = \
(updatedCSV2_IS_StayRangeInfo_IS_StartTime
.loc[mask, '当月抗生素使用时间区间']
.apply(calCurrentMonthAntiBioticsUseDays)
)
updatedCSV2_IS_StayRangeInfo_IS_StartTime['使用次数'] = \
(updatedCSV2_IS_StayRangeInfo_IS_StartTime.使用天数 * updatedCSV2_IS_StayRangeInfo_IS_StartTime.频次_int).round(0) # 四舍五入到整数
# 与ddd字典拼接
DDDmetrics = pd.read_csv('dict/ddd测算字典.csv')
# 判断字典是否存在重复的行,以正确merge
duplicate_mask = DDDmetrics.duplicated(subset=['抗生素名称'], keep=False)
# 输出重复的行
if duplicate_mask.any():
print("\n存在重复的“抗生素名称”:")
print(DDDmetrics[duplicate_mask])
else:
print("不存在重复的“抗生素名称”。")
updatedCSV2_IS_StayRangeInfo_IS_StartTime_M = \
updatedCSV2_IS_StayRangeInfo_IS_StartTime.merge(DDDmetrics, how='left') # merge后这里少行了,故用how='left'方法,保证不少行,外键为"抗生素名称"
updatedCSV2_IS_StayRangeInfo_IS_StartTime_M['抗生素ddds'] = (updatedCSV2_IS_StayRangeInfo_IS_StartTime_M['使用次数'].astype(float) \
* updatedCSV2_IS_StayRangeInfo_IS_StartTime_M['用量'].astype(float) \
* updatedCSV2_IS_StayRangeInfo_IS_StartTime_M['转换系数'].astype(float) \
/ updatedCSV2_IS_StayRangeInfo_IS_StartTime_M['DDD'].astype(float)).round(2)
# 删除交长嘱使用,和使用天数小于0的行,
# updatedCSV2_IS_StayRangeInfo_IS_StartTime_M = \
# updatedCSV2_IS_StayRangeInfo_IS_StartTime_M[(updatedCSV2_IS_StayRangeInfo_IS_StartTime_M.备注 != '交长嘱使用') \
# & (updatedCSV2_IS_StayRangeInfo_IS_StartTime_M.抗生素ddds>=0)]
# 处理没有开抗生素时间的,很简单,ddds为0
updatedCSV2_IS_StayRangeInfo_Not_StartTime['抗生素ddds'] = 0
# 有无开嘱时间的两部分合并,完成有转科信息部分的汇总
updatedCSV2_IS_StayRangeInfo_M = pd.concat([updatedCSV2_IS_StayRangeInfo_IS_StartTime_M, updatedCSV2_IS_StayRangeInfo_Not_StartTime], ignore_index=True)
###### 处理无转科信息病人的抗生素使用情况,
# 分两种情况:有开抗生素医嘱时间和无开抗生素时间
# 先去重,减小计算量,不影响正确结果,
updatedCSV2_Not_StayRangeInfo.drop_duplicates(subset=['科室名称', '住院号', '抗生素名称', '开抗生素时间'], inplace=True)
# 将字符串类型的时间转换成 Timestamp 类型,后面有去重和计算要小心类型不一致
updatedCSV2_Not_StayRangeInfo['开抗生素时间'] = pd.to_datetime(updatedCSV2_Not_StayRangeInfo['开抗生素时间'])
updatedCSV2_Not_StayRangeInfo_IS_StartTime, updatedCSV2_Not_StayRangeInfo_Not_StartTime = updatedCSV2_Not_StayRangeInfo.query('开抗生素时间.notnull()').copy(), updatedCSV2_Not_StayRangeInfo.query('开抗生素时间.isnull()').copy()
# 处理临嘱,判断开抗生素时间是否为当月
updatedCSV2_Not_StayRangeInfo_IS_StartTime.loc[updatedCSV2_Not_StayRangeInfo_IS_StartTime.医嘱类型=='临嘱', '是否当月住科期间内'] = \
updatedCSV2_Not_StayRangeInfo_IS_StartTime.apply(lambda row : is_within_CurrentMonth_interval(row, year=year, month=month, last_day=last_day), axis=1)
updatedCSV2_Not_StayRangeInfo_IS_StartTime['当月内有效医嘱'] = updatedCSV2_Not_StayRangeInfo_IS_StartTime['是否当月住科期间内']
#处理长嘱,相对复杂,但比有转科信息的病人明显简单
### 注意,停抗生素时间的空字符不是空值,用fillna处理不了,因此先将''替换成np.nan,深坑二
# updatedCSV2_Not_StayRangeInfo_IS_StartTime.loc[updatedCSV2_Not_StayRangeInfo_IS_StartTime.医嘱类型=='长嘱', '停抗生素时间'] = \
# updatedCSV2_Not_StayRangeInfo_IS_StartTime.loc[updatedCSV2_Not_StayRangeInfo_IS_StartTime.医嘱类型=='长嘱', '停抗生素时间'].replace('', np.nan)
# 将同一组的行打上相同的标签,标签作为新的一列加入 DataFrame
updatedCSV2_Not_StayRangeInfo_IS_StartTime.loc[updatedCSV2_Not_StayRangeInfo_IS_StartTime.医嘱类型=='长嘱', '长嘱组标签']= \
updatedCSV2_Not_StayRangeInfo_IS_StartTime[updatedCSV2_Not_StayRangeInfo_IS_StartTime.医嘱类型=='长嘱']\
.groupby(['住院号', '抗生素名称', '开抗生素时间']).ngroup().apply(lambda x: f'-{x}')
# # 将同一组的抗生素时间双向填充,得到实际的长嘱停嘱时间
# updatedCSV2_Not_StayRangeInfo_IS_StartTime.loc[updatedCSV2_Not_StayRangeInfo_IS_StartTime.医嘱类型=='长嘱', '停抗生素时间'] = (
# updatedCSV2_Not_StayRangeInfo_IS_StartTime[updatedCSV2_Not_StayRangeInfo_IS_StartTime.医嘱类型=='长嘱']
# .groupby(['住院号', '抗生素名称', '开抗生素时间'])['停抗生素时间']
# .apply(lambda x: x.fillna(method = 'ffill').fillna(method = 'bfill')) # 组内双向填充,不会漏
# )
# 剩下为空值的停抗生素时间为未停的抗生素,统一设置为当天晚上的23:59:59
# updatedCSV2_Not_StayRangeInfo_IS_StartTime.loc[
# (updatedCSV2_Not_StayRangeInfo_IS_StartTime.医嘱类型=='长嘱') & (updatedCSV2_Not_StayRangeInfo_IS_StartTime['停抗生素时间'].isnull()),
# '停抗生素时间'] = pd.Timestamp(currentDateTime)
# 计算开抗生素时间和停抗生素时间在住科期间的叠加区间,此时等同于开抗生素时间和停抗生素时间的区间,不会产生空字符
mask = updatedCSV2_Not_StayRangeInfo_IS_StartTime.医嘱类型 == '长嘱'
columns = ['开抗生素时间', '停抗生素时间']
updatedCSV2_Not_StayRangeInfo_IS_StartTime.loc[mask, '住科期间抗生素使用时间区间'] = \
updatedCSV2_Not_StayRangeInfo_IS_StartTime.loc[mask, columns].apply(
lambda x: f"{x[0]}~{x[1]}" if pd.notnull(x[0]) and pd.notnull(x[1]) else np.nan,
axis=1)
# 计算长嘱的当月抗生素使用时间区间,#注意,会产生空字符(函数返回的)
updatedCSV2_Not_StayRangeInfo_IS_StartTime.loc[mask,
'当月抗生素使用时间区间'] = \
updatedCSV2_Not_StayRangeInfo_IS_StartTime.loc[mask,
'住科期间抗生素使用时间区间']\
.apply(lambda x: getCurrentMonthStayRange(x, year=year, month=month, last_day=last_day))
# # '当月抗生素使用时间区间'为空字符的,'是否当月住科期间内'为False,不为空字符则为True
updatedCSV2_Not_StayRangeInfo_IS_StartTime.loc[mask, '是否当月住科期间内'] = \
~updatedCSV2_Not_StayRangeInfo_IS_StartTime.loc[mask, '住科期间抗生素使用时间区间'].eq('')
updatedCSV2_Not_StayRangeInfo_IS_StartTime.loc[
mask, '当月内有效医嘱'] = updatedCSV2_Not_StayRangeInfo_IS_StartTime.loc[
mask, '是否当月住科期间内']
# 筛选有效住科期间的数据
updatedCSV2_Not_StayRangeInfo_IS_StartTime = updatedCSV2_Not_StayRangeInfo_IS_StartTime.query('当月内有效医嘱==True')
# # 计算抗生素使用强度
# 计算使用频次
updatedCSV2_Not_StayRangeInfo_IS_StartTime['频次_int'] = updatedCSV2_Not_StayRangeInfo_IS_StartTime.频次.apply(freq)
# 计算使用天数,临嘱为1天
updatedCSV2_Not_StayRangeInfo_IS_StartTime.loc[updatedCSV2_Not_StayRangeInfo_IS_StartTime['医嘱类型']=='临嘱', '使用天数'] = 1
# 计算使用天数,长嘱按“当月抗生素使用时间区间”计算,可能有多个区间。
updatedCSV2_Not_StayRangeInfo_IS_StartTime.loc[
mask, '使用天数'] = \
(updatedCSV2_Not_StayRangeInfo_IS_StartTime
.loc[mask, '当月抗生素使用时间区间']
.apply(calCurrentMonthAntiBioticsUseDays)
)
updatedCSV2_Not_StayRangeInfo_IS_StartTime['使用次数'] = \
(updatedCSV2_Not_StayRangeInfo_IS_StartTime.使用天数 * updatedCSV2_Not_StayRangeInfo_IS_StartTime.频次_int).round(0) # 四舍五入到整数
# 与ddd字典拼接
updatedCSV2_Not_StayRangeInfo_IS_StartTime_M = \
updatedCSV2_Not_StayRangeInfo_IS_StartTime.merge(DDDmetrics, how='left') # merge后这里少行了,故用how='left'方法,保证不少行,外键为"抗生素名称"
updatedCSV2_Not_StayRangeInfo_IS_StartTime_M['抗生素ddds'] = (updatedCSV2_Not_StayRangeInfo_IS_StartTime_M['使用次数'].astype(float) \
* updatedCSV2_Not_StayRangeInfo_IS_StartTime_M['用量'].astype(float) \
* updatedCSV2_Not_StayRangeInfo_IS_StartTime_M['转换系数'].astype(float) \
/ updatedCSV2_Not_StayRangeInfo_IS_StartTime_M['DDD'].astype(float)).round(2)
# 删除交长嘱使用,和使用天数小于0的行,
# updatedCSV2_Not_StayRangeInfo_IS_StartTime_M = \
# updatedCSV2_Not_StayRangeInfo_IS_StartTime_M[(updatedCSV2_Not_StayRangeInfo_IS_StartTime_M.备注 != '交长嘱使用') \
# & (updatedCSV2_Not_StayRangeInfo_IS_StartTime_M.抗生素ddds>=0)]
# 处理没有开抗生素时间的,很简单,ddds为0
updatedCSV2_Not_StayRangeInfo_Not_StartTime['抗生素ddds'] = 0
# 有无开嘱时间的两部分合并,完成无转科信息部分的汇总
updatedCSV2_Not_StayRangeInfo_M = pd.concat([updatedCSV2_Not_StayRangeInfo_IS_StartTime_M, updatedCSV2_Not_StayRangeInfo_Not_StartTime], ignore_index=True)
# 合并有、无转科信息的两部分组成全院数据
updatedCSV2Hosp = pd.concat([updatedCSV2_IS_StayRangeInfo_M, updatedCSV2_Not_StayRangeInfo_M], ignore_index=True)
# 统一处理临嘱有停抗生素时间的行,抗生素ddds设为0,
updatedCSV2Hosp.loc[(updatedCSV2Hosp.医嘱类型=='临嘱') & (updatedCSV2Hosp.停抗生素时间.notnull()), '抗生素ddds'] = 0
updatedCSV2Hosp.loc[(updatedCSV2Hosp.医嘱类型=='临嘱') & (updatedCSV2Hosp.停抗生素时间.notnull()), '使用次数'] = 0
# 数据汇总
# 前端字段
frontEndCols = ['科室名称', '住院号', '病人姓名', '床号', '管床医生', '医嘱类型', '抗生素名称', '用量',
'单位', '频次', '开抗生素时间', '停抗生素时间','医嘱日期', '计算日期区间的住院天数','使用天数','DDD', '转换系数', '抗生素ddds']
deptData = updatedCSV2Hosp[frontEndCols]
controlLineDict = pd.read_csv('dict/各科室ddd控制指标.csv')
month = str(month).zfill(2)
day = str(day).zfill(2)
_=[]
for dept in deptData.科室名称.unique():
deptDF = deptData.query('科室名称==@dept').sort_values(by=['床号', '住院号'])
deptDF['病人ddds'] = deptDF.groupby('住院号').抗生素ddds.transform('sum').round(2)
deptDF['抗生素使用强度'] = (100 * deptDF.病人ddds / deptDF.计算日期区间的住院天数).round(1)
deptDF.to_csv(f'data/wrangledAntibioMedcalOrders/科室/{dept}-{year}年{month}月.csv', index=False)
deptStayDays = deptDF.groupby('住院号').计算日期区间的住院天数.agg('mean').sum()
deptDDDs = deptDF.groupby('住院号').病人ddds.agg('mean').sum().round(2)
if deptStayDays != 0 :
deptIntensity = (100 * deptDDDs / deptStayDays).round(1)
else:
deptIntensity = np.nan
controlLine = controlLineDict.loc[controlLineDict.科室==dept,'指标']
if not controlLine.empty:
excess = (deptIntensity - controlLine.values[0]).round(1)
else:
excess = np.nan
_.append([year, month, day, dept, deptDDDs, deptStayDays, deptIntensity, excess])
_ = pd.DataFrame(_,columns=['年','月', '日', '科室名称', 'ddds','住院天数','使用强度','超标'])
_f = f'data/wrangledAntibioMedcalOrders/全院/科室汇总-{year}年{month}月.csv'
if not os.path.exists(_f):
_.to_csv(_f, index=False)
else:
_e = pd.read_csv(_f, dtype={'年':str, '月':str, '日':str})
pd.concat([_e, _], ignore_index=True).drop_duplicates(subset = ['年','月', '日', '科室名称'], keep='last').to_csv(_f, index=False)
_i = (100 * _.ddds.sum()/_.住院天数.sum()).round(1)
_a = pd.DataFrame([[year, month, day, _.ddds.sum().round(2), _.住院天数.sum(), _i, (_i - 40).round(1)]], columns=['年','月', '日', 'ddds','住院天数','使用强度','超标'])
_f1 = f'data/wrangledAntibioMedcalOrders/全院/全院汇总-{year}年{month}月.csv'
if not os.path.exists(_f1):
_a.to_csv(_f1, index=False)
else:
_e1 = pd.read_csv(_f1, dtype={'年':str, '月':str, '日':str})
pd.concat([_e1, _a], ignore_index=True).drop_duplicates(subset = ['年','月', '日'], keep='last').to_csv(_f1, index=False)
return None